Microambientes, pH, control redox, bioenergética y fenotipos intermedios
Warning: Undefined array key "file" in /home/bioacylc/domains/bioacyl.com/pass.bioacyl.com/wordpress/wp-includes/media.php on line 1788
Warning: Undefined array key "file" in /home/bioacylc/domains/bioacyl.com/pass.bioacyl.com/wordpress/wp-includes/media.php on line 1788
¿Cómo lo inmaterial se convirtió en vida?
La vida dio inicio cuando proto-células en fuentes hidrotermales alcalinas oceánicas, dominaron la producción y acumulación de energía por transporte de electrones en cadena, apartando gradientes iónicos al través de proto-membranas suficientemente estañas y así lograr independizarse de los gradientes geoquímicos provistos por las fuentes. Lo anterior implicó la creación de un microambiente con un flujo de energía suficientemente alta (entalpía) que mantuviera un estado de entropía bajo. El estado de entropía bajo de los células vivientes, requiere de un decremento ulterior del flujo entálpico para mantener un intercambio negativo de energía libre(ΔG = ΔH – TΔS), solamente sostenible bajo provisión constante de alta energía. Pero al generar, y lograr conservar su propia bioenergía, las proto-células pudieron desvincularse de las fuentes y volverse autónomas. Si bien universalmente, las únicas formas de proveerse de bioenergética apta para fijar carbono, crear biomasa y prosperar como seres autónomos, lo es primero por acoplamiento de la sintasa ATP a la proto-membrana mediante y la captura de la energía liberada durante la respiración celular por oxidación fosforilativa, y en segundo lugar la fosforilación de substratos externos para la conversión de la ADP en ATP, durante la catálisis de una reacción fuertemente exergónica y gradientes dependientes en la bifurcación de electrones basada en flavina, en tercer lugar (Sousa, Thiergart, Landan, Nelson-Sathi, Pereira, Allen, Lane, & Martin, 2013). Pero al evolucionar, las células lograron no solamente almacenar suficiente energía mediante gradientes iónicos (quimiosmótico=fuerza proto-motriz) al través de membranas, pero también del mismo proceso, reutilizar el potencial eléctrico del espacio intermembranoso para convertir la ADP en ATP, y la reconversión de la energía eléctrica a energía química incorporando la generación de gradiente de protones basada en quinonas y la conversión por fosforilación de substratos, adaptada al suministro interno de substratos. Este mecanismo bioenergético ‒tan universal como el código genético (Lane & Martin, 2012)‒ que permite la conservación de energía, se ajustó de maravilla a la complejidad que es inmanente a la vida de seres complejos dotados de funciones multidimensionales. El intercambio (Redox) logra sincronizar y armonizar, en el seno de un sistema biológico multiestratificado, desde el microambiente celular, al intercelular y el intercambio interno en órganos y finalmente, integrar el metabolismo a nivel organísmico. No solamente, está también dotado de una faceta adaptativa, la cual responde a variaciones de O2, la luz, oscuridad y espacio-temporales (ritmos circadianos). Además se adapta a estímulos estresantes externos. Es decir, la química redox es versátil y además de ser apta para darle cabida a modificaciones estructurales biológicas, asimismo responde a cambios ambientales (Jones & Sies, 2015). Son modificaciones que, además de estructurar membranas, conducen al desarrollo de vesículas, vacuolas y organelas en los seres evolucionados. Es la bioquímica de los seres vivos y muy diferente a la química de los objetos inanimados; si bien ambas utilizan muchos elementos en común, incluyendo en el lado biológico hasta metales e inorgánicos, como Fe, Cu, Zn, Mb, Mn, Tn, Se.
El gradiente quimiosmótico parece haber nacido, al haberse configurado el ancestro eucariótico común de una simbiosis entre un hospedero estricto-dependiente en hidrógeno (archaebacterium) y un simbionte productor y liberador de hidrógeno como producto de desecho de metabolismo anaeróbico heterotrófico (eubacteriun), estrechamente ligado a hidrógeno. Esta teoría de (Martin & Müller, 1998) es congruente con los postulados de la teoría quimiosmótica P. Mitchell, por cuanto esta asume que el gradiente se acumula en el espacio intermembranoso de las mitocondrias, alimentado por la fosforilación oxidativa dependiente de la síntesis de ATP la cual bombea los protones desde la membrana interna en la matriz, al espacio referido. Cuatro protones son extruidos por los complejos I y IV y 2 por el complejo II de la cadena respiratoria transportadora de electrones y luego en la catalización por la sintasa ATP de ADP a ATP, 3 protones son devueltos a la matriz: De manera que se forma un ΔpH de microambiente alcalino en la matriz y otro ácido en el espacio intermembranoso. El aumento del diferencial es conducente a mayor producción de ATP. Cuando las membranas evolucionaron a no permeables, el bombeo de protones emergió y acompañado necesariamente por energética de antiportadores de Na+, mecanismo que permitió mantener el gradiente quimiosmótico y evitar la estabilización iónica del citosol con el ambiente, gracias al desarrollo de un antiportador capaz de transducir el gradiente geoquímico (H+) en uno bioquímico (Na+), y que selectivamente expulsara Na+ del citosol hacia el exterior. Lo anterior junto con la hiperpolarización de la membrana, el mantenimiento del pHi y el balance extra/intracelular de iones y protones. Precisamente esta segregación en compartimentos y las diferencias de potencial que se generan entre ellos, son las características que les permite a los eucariotas, proveerse autónomamente de la energía necesaria para el desenvolvimiento, señalización y el mantenimiento de la vida.
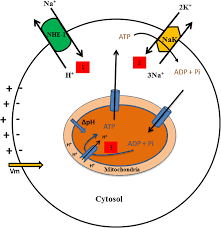
Entrelazamiento entre ATP/ADP, pHi y el potencial de membrana Vm. El intercambiador sodio-protón (NHE-1) extruye un protón en contra de un ión de sodio (Na+). El intercambiador electrogénico sodio-potasio (NaK-ATPase), consumidor importante de ATP en eucariotas, extruye 3 sodios (Na+) del citosol contra 2 potasios (K+) que ingresan al citosol y crea la hiperpolarización de la membrana celular. El ΔpHi entre la matriz de la mitocondria (ver texto) y el espacio intermembranoso, mediante la fuerza proto-motriz, impulsa la síntesis de ATP.
La presencia de un ciclo redox en el seno del ciclo celular y la marcada susceptibilidad al pHi en cada punto de verificación, que permite el avance del proceso a cada fase sucesiva, señala que un determinado microambiente vigila que paulatinamente se hayan cumplido las condiciones aptas para la progresión, hacia la mitosis. 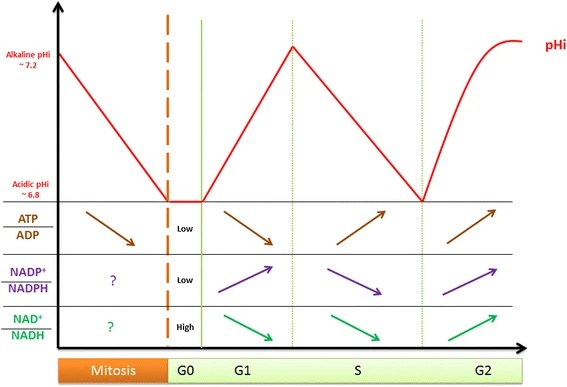
La mitosis es comunmente descrita como un evento catastrófico, donde para satisfacer la demanda energética, la despolimerización microtubular y el operar de las bombas ATP-dependientes, queman el ATP por hidrólisis. Eso disminuye la relación ATP/ADP. Al mismo tiempo el pHi disminuye y se estanca por su periodo más extenso en células inactivas, fase G0. No existen datos de la relación redox NAD+/NADH y NADP+/NADPH durante la mitosis. En la fase G1 temprana, el aumento de glicólisis refleja transitoriamente un pHi ácido antes de que el citosol se alcalinice. Se asume que esto es debido a síntesis de lactato. La enzima lactato dehidrogenasa cataliza la conversión a ácido láctico mediante la utilización de NADH como coenzima. La relación NAD+/NADH es alta en G1 y disminuye luego, para alcanzar su valor mínimo duratnte la fase S. La necesidad de síntesis de ácidos nucléicos durante esta fase y el consumo de NADPH para la reducción a especies ROS-reducidas, induce la entrada al ciclo celular y aumenta el sendero de fosfato de pentosa (PPP), mediante aumento de NADP+/NADPH en la fase S. Durante esta fase la acidificación del citosol, probablemente debida a glutaminolisis, se topa con mitocondrias en estado de hiperperfusión durante síntesis de ATP. La síntesis de histonas que depende de NAD+, tiende a dismunuir la NAD+/NADH. En la fase G2, sea la ATP y el pHi alcanzan su nivel máximo y el consumo de NADPH durante la síntesis de ácidos grasos, aumenta la NADP+/NADPH. Lo anterior es debido al aumento de transbordamiento de malato/citrato, lo que permite la síntesis de NAD+, por la conversion de oxalacetato a malato.
De lo expuesto resulta evidente que la química que da forma a seres autónomos los cuales desafían las leyes de la termodinámica y que operan sistemas abiertos y estables lejos de equilibrio, son dependientes de ese potencial quimiosmótico y que el mantenimiento de un nivel mínimo del potencial, es capital para su sobrevivencia. De hecho, si el potencial disminuye por debajo de un cierto nivel, la célula arriesga de entrar en el proceso de necrosis. Mientras que para la muerte programada o apoptosis, el nivel intracelular de ATP se sostiene relativamente alto. Lo último resulta lógico, visto y considerado que, la apoptosis no entra en juego en presencia de daño celular o estrés únicamente, sino que interviene como parte de la regulación del desarrollo y la morfogénesis normal (el no excedente de células en la formación) (Nikoletopoulou, Markaki, Palikaras, & Tavernarakis, 2013).
Ha sido corroborado mediante experimentos (Ataullakhanov & Vitvitsky, 2002), (Özalp, Pedersen, Nielsen, & Olsen, 2010), (Ytting, Fuglsang, Kalervo Hiltunen, Kastaniotis, Özalp, Nielsen, & Olsen, 2012) que, funcionalmente es más importante la relación ATP, ADP y AMP que no la concentración absoluta de ATP. La «carga energética» del sistema de adenilato, definida como AEC = ([ATP] +0.5[ADP])/([ATP] + [ADP] + [AMP]), es un parámetro básico que refleja el estado buferizado de las reservas energéticas de las células. Propuesto por Atkinson en 1967, es un índice en escala entre 0 y 1. Cuando la charca de nucleótidos es en la forma de AMP, el AEC es cero y el sistema está descargado completamente (concentración 0 de ATP y ADP). Cuando es solo de ADP, la carga es 0.5. Si toda la charca de nucleótidos fuera en ATP, la carga es 1. Las enzimas que utilizan ATP, son inhibidas por ADP o AMP, respondien con con curvas de alta pendiente en velocidad según la carga, igual como responden enzimas reguladas por substrato y sin sitios múltiples de enlace cooperativo.
Desde la primera corroboración experimental de la ecuación, ha sido evidente que múltiples organimos en condiciones óptimas de crecimiento mantienen valores AEC, entre AEC=0.7 y AEC=0.95, en muchos casos con estabilización en 0.9. Como se dijo, este valor es el resultado del poder búfer de la triada ATP, ADP y AMP (o la alternativa de cada proceso en usar GTP, UTP, TTP o CTP selectivamente e individualmente variando la charca de nucleótido trifosfatado) ya que se observan fluctuaciones marcadas en la concentración de cada nucleótido separadamente. Atkinson y coautores, concluyeron que para los valores del AEC referidos, la mayoría de la reacciones productoras de ATP se encuentran en balance con las que son counsumidoras de ATP. En condiciones desfavorables si el AEC decae por debajo de los valores determinados, se induce la muerte celular (Atkinson & Walton, 1967)(Atkinson & Walton, n.d.).
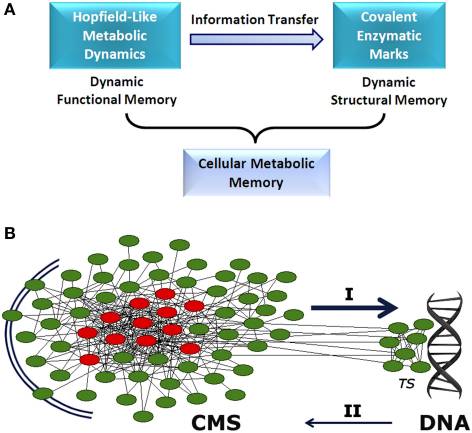
Memoria metabólica celular.
(A) Dinámica tipo atractivo–Hopfield-simil puede emerger en las redes metabólicas. Tienen la capacidad de almacenar los patrones catalíticos funcionales y que posteriormente pueden ser accesados ante un estímulo apropiado. La dinámica metabólica almacenada es estable y funge como memoria a largo plazo. La información específica puede ser tranferida a los procesos enzimáticos pos-translacionales y la modulación covalente de los mismos. De manera que la información funcional almacenada y derivada de procesos catalíticos, puede ser luego incrustada para moldear marcas moleculares estructurales. (B) Entre el ambiente extracelular y el DNA, la estructura metabólica celular (CMS), un sistema de procesamiento descentralizado de información con capacidad de almacenar memoria metabólica, establece un intercambio informático entre estas. El CMS genera periféricamente instrucciones bioquímicas en manojos, que modulan a cada actividad enzimática en particular según las señales externas que percibe para auto-regularse y, simultáneamente adaptarse al medioambiente externo. Envía adicionalmente y hacia el núcleo, un flujo de señales moleculares a la red metabólica asociada al DNA, el cual gobierna el sistema de información transcripcional. Esos flujos permiten precisión en la expresión genética y síntesis de polipéptidos requeridos para el mantenimiento del brazo adaptativo del CMS. Ambos sistemas de información, se coordinan para modular el desarrollo fisiológico celular. I: flujo regulatorio del sistema transcripcional. II: reposición de polipéptidos. TS: sistema transcripcional. La red CMS está representada: Círculos rojos: meollo metabólico estable; Círculos verdes; procesos intermitentes de encendido/apagado. (De la Fuente, 2015).
Definido en términos generales, el metabolismo es la suma de procesos bioquímicos en los seres vivientes que produce o consume energía. La cuantía de esos procesos es gigantesca y consta de 8700 reacciones y 16000 metabolitos; los cuales se encuentran anotados en la biblioteca de Genes y Genomas de Kyoto (http://www.genome.jp/kegg/pathway.html). Puede describirse de manera simplificada, como el conjunto de nutrientes abundantes, tales como carbohidratos, ácidos grasos y amino ácidos, que son esenciales para la homeoestasis energética y la síntesis macromolecular en humanos. Funcionalmente puede se dividido en 3 clases: las reacciones que sintetizan moléculas simples y las polimerizan en macromoléculas complejas (anabolismo); las que degradan las macromóculas para liberar energía (catabolismo); y las que ayudan a eliminar residuos tóxicos producidos por las dos primeras clases. Es vital, en vista de que provee la fuente de energía que le permite a los seres vivientes resistir el ímpetu termodinámico de degradar en entropía y mantenerse lejos de equilibrio, lo que les permite conservar y estabilizar así, su vitalidad (DeBerardinis & Thompson, 2012).
La integración de senderos bioquímicos en la respuesta celular a los factores de crecimiento, es un buen ejemplo de lo anterior. Los factores de crecimiento estimulan la transducción del sendero fosfoinositol 3’cinasa (PI3K)/Akt/Diana de rapamicina en células de mamífero. La activación de este sendero y otros, altera el estado de fosforliación de elementos que en conjunto coordinan las funciones celulares que culminan en la división celular. Pero una transición exitosa de estado de reposo a crecimiento celular, ocurre solamente si el metabolismo es reprogramado para abastecer la demanda aumentada que la proliferación celular demanda. De manera que el estado metabólico, debe de econtrarse en estado permisivo para el crecimiento celular (Lum, Bauer, Kong, Harris, Li, Lindsten, & Thompson, 2005). En el caso específico de PI3K/Akt/mTOR, se estimula una capatación rápida de nutrientes esenciales y su respectiva asignación anabólica y catabólica, para contar con la energía que conlleve a la formación de macromoléculas (Gibbons, Abraham, & Yu, 2009). La interrupción de dicha captación, torna al factor de crecimiento inefectivo.
De manera que la noción hasta ahora aceptada, en cuanto a que el estado regulatorio celular guiado por factores transcripcionales predomina sobre el estado metabólico, cambia visto desde la perspectiva de biología de sistemas, de los conceptos de bioenergéticca y de flujo de información; al dar debida cuenta del retorno de retroinformación que el estado metabólico genera y perfectamente apta, de gravitar recíprocamente sobre la transcripción. La apreciación de esta reciprocidad, es necesaria para comprender muchos aspectos de investigación biológica hasta ahora insolutos. No es aceptable seguir considerando el metabolismo como un proceso auto-regulado e independiente de los otros sistemas biológicos. Más bien, el metabolismo impacta, o es virtualmente impactado por cada otro proceso molecular. No es de sorprenderse, los aspectos fundamentales del metabolismo energético están altamente conservados y anteceden a los aspectos relacionados con la organización compleja de los seres muticelulares; organización que surgió mucho tiempo después de los procesos fundamentales. Es más, no sería posible la estructuración molecular sin el flujo de información funcional derivado de procesos catalíticos, estructuración sobre la cual se fundamenta la organización mayor y la entrada en escena de la transcripción y la transducción; caraterística en los seres multicelulares en estado de homeoestasis.
La estructura metabólica celular (CMS) y estado del microambiente
Seguir en el enfoque de tratar de hallar intervenciones que corrijan disfuncionalidades de seres formados por 3.72 × 1013 de unidades celulares que dependen de un tenor bioenergético primordial para mantenerse vitales y que además, constituyen redes de comunicación para transmitir y procesar información entre ellas que les permite tomar decisiones, no puede más que llevar a resultados parciales y fallidos cuando se destima esa realidad. Si se considera que en dicha vitalidad interviene un flujo informático metabólico primordial como medio de comunicación y el cual impacta recíprocamenteal núcleo y los genes de cada célula, es evidente que dichos factores deben de ser tomados en cuenta para diseñar intervenciones que restablezcan el estado previo y no limitarse a señalizaciones de ligando-receptor únicamente, sin tomar en cuenta que dicha información necesita de un lugar propicio (topología del microambiente) para que fluya. Al flujo de información metabólico primordial hay que agregar, el estímulo de la misma célula autócrino, la comunicación física entre células, estímulos secretados de células cercanas o lejanas de órganos distintos, factores protéicos secretados como factores de crecimiento, citocinas (West, McCuaig, Franchini, & Powrie, 2015), quimiocinas, canales iónicos, metabolitos (Lyssiotis & Kimmelman, 2017) y vesículas extracelulares (Becker, Thakur, Weiss, Kim, Peinado, & Lyden, 2016), sin olvidarse de la condición fisiológica del microambiente, como las condiciones físicas y bioquímicas de la matriz extracelular, el pH, el He, el nivel de oxígeno (hipoxia) y de los nutrientes (deprivación energética). Son todos factores a los cuales las células responden para inducir el comportamiento celular congruente con el cumplimiento de las funciones biológicas complejas, que son propias de los tejidos. El conjunto de las interacciones funcionales relevantes que permiten la distribución de dicha información a multi-escala dinámica en tejidos humanos puede ser definida como «interactoma».
Para visualizar mediante herramientas matemáticas la complejidad del interactoma y conocer los fenotipos que a la postre se manifiesta en los individuos, con el fin de relacionar los procesos subyacentes y sus disfunciones y sacar deducciones útiles, las simplificaciones que caracterizan a la ciencia de redes, han constitiudo la manera práctica de ver con mayor claridad dentro del contexto de una complejidad que de otra manera, es abrumadora. El progreso científico de las últimas 2 décadas basado sobre estudios genéticos (análisis de vinculación y GWAS(genome-wide association studies), de las plataformas de cribado de alto redimiento—análisis OMICS (secuenciación genómica, análisis de expresión transcripcional, análisis proteinómicos, etc), de estudios bioquímicos (metabalomicos), junto con datos moleculares, celulares, fisiológicos y clínicos han puesto al alcance la información necesaria que permite diseñar tales simplicaciones. No obstante, el cuerpo de datos comprende: ~25000 genes encodificadores de proteínas, ~1000 metabolitos, y un número creciente de proteinas modificadas postranslacionalmente, molélucas funcionales de ARN, que en conjunto exceden 100000 participantes más el hecho de que funcionalmente algunos elementos se ubican superpuestos sobre otros generando planos multidimensionales (Barabasi, Gulbahce, & Loscalzo, 2011).
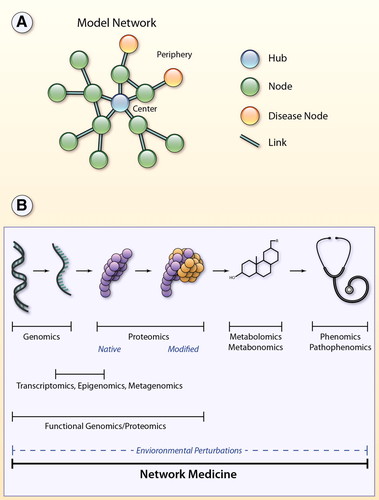
Metabonomics=Medición cuantitativa de la respuesta y adaptación metabólica multiparamétrica , de un sistema (multiceluar) complejo hacia una perturbación fisiopatológica o una modificación genética en el tiempo.
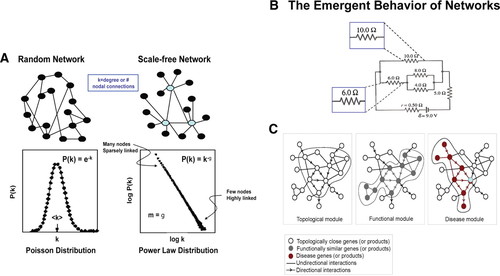
Las redes se utilizan para representar una amplia variedad de sistemas biológicos. En su forma más sencilla una red puede ser representada mapeando un conjunto de entidades moleculares, llamados «nodos» y los cuales se posicionan de acuerdo a sus interconexiones funcionales entre los nodos, o «conexiones». En medicina de redes, un nodo puede representar un factor biológico (i.e., gen, cromosoma, metabolito, ARN no.codificado o hasta un patofenotipo). Una conexión puede representar interacciones físicas, inducción transcriptómica, activación enzimática, conversión de un metabolito a otro, o hasta genes/rasgos compartidos en las manifestaciones de enfermedad. Las interacciones no ocurren en forma aleatoria en humanos y las redes consisten de una minoría de nodos altamente inteconectados conotros nodos (centro de actividad=hub), que son esenciales para mantener la integridad de la red y las funciones celulares esenciales, y de una minoría de nodos periféricos escasemente conectados. Generalmente los genes de las enfermedades complejas y crónicas no transmisibles están típicamente localizados en al periferia de la red, con escasas conexiones en comparación a los centros de actividad (Barabasi et al., 2011; Albert, Jeong, & Barabasi, 2000). Algunos centros de actividad se conectan con otros enracimamientos de nodos para preservar y mantener la integridad del todo. Son redes sin escala, con probabilidad de que el número de las conexiones por nodo siga una distribución (P(k)=k−γ de potencia recibida por una ley log-normal donde=(k es el n° e interacciones) (y es la pendiente de la trama log P(k) vs log(k))Fig A (Barabasi & Albert, 1999; Albert & Barabasi, 2002). Las distribuciones por ley de poder disminuyen más lentamente que las distribuciones de redes aleatorias, en que las interconexiones entre nodos son fortuitas; cada nodo con igual probabilidad de conectarse a cualquier otros nodo del sistema. La probabilidad resultante de redes aleatorias, P, de conexiones por nodo, sigue la distribución tipo Poisson, Fig A (Loscalzo & Barabasi, 2011; Barabasi et al., 2011). Por esa razón las redes sin escala pueden definirse como «sobredeterminadas» de donde se infiere que para comprender el funcionamiento a nivel de sistema no es necesario definir todos los componentes en su totalidad. Se trata de comportamientos emergentes, de tal manera que el todo no es la simple suma de sus partes y la manifestación fenotipica (rol in vivo), menos aún puede extrapoladarse del íntimo conocimiento de un solo nodo. Fig B.
Las redes sin escala facilitan la diversidad química con expendio mínimo de energía, asi como
la transción entre estados metabólicos (Wagner & Fell, 2001). Son paradigmáticas de la selección natural y la evolución: en redes complejas de genes, las mutaciones o eliminación de un gen ubicado en un centro de actividad, resulta en mortalidad embrionaria, mientras que las mutaciones de genes periféricos débilmente conectados, permite la diversidad biológica y la selección natural (Oikonomou & Cluzel, 2006). Impulsado por mutación aleatoria y selección, las redes sin escala son capaces de evolucionar rápidamente hacia un estado funcional óptimo, sin necesidad de afinamiento (Albert et al., 2000). Las redes minimizan las consecuencias de la mayoría de los errores bioquímicos o errores genéticos (Wagner & Fell, 2001), y modulan las pertubación que puedan ocasionar los errores para que los efectos sean mínimos en las funciones críticas (Pastor-Satorras & Vespignani, 2002): excepto que los centros de actividad sean el foco de los errores. La naturaleza sobrederminada permte la introespección en la función de la red incluso cuando elementos de la red pueden estar ausentes (o el conocimiento de la red sea incompleto),
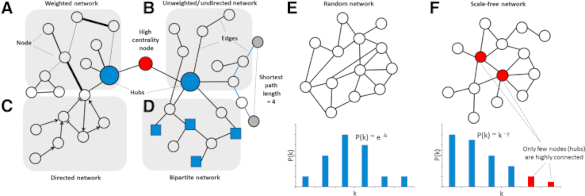
Las relaciones entre nodos se representan por líneas conectoras que representan las conexiones y corresponden a interacciones moleculares, tales como la regulación de genes, interacción física interproteica, o metabolismo de un substrato Fig. Grados se refiere al número de conexiones de un nodo. Nodos de alto grado son aquellos que pueden ser identificados de alguna entidad biológica que desempeña un rol central dentro de la red. (grado de centralidad). La intensidad de la interacción puede ser ponderada (grosor de la conexión) o no ponderada. Pueden ser direccionadas (flechas) or no direccionadas. Utilizando la teoría de gráficos las relaciones descritas pueden ser visualizadas y analizadas. La conexión entre 2 nodos se logra a veces mediante otros nodos y el mínimo de conexiones para lograr la interacción se considera un sendero. El grado en ese caso del ejemplo de fig = 4.
Propiedades de redes y tipos.
Los nodos rewpresentan emtidades biológicas específicas y en contacto con otros mediante conexiones. Los centros de actividad, altamente conectados, frecuentemente representas a wlwmentos biológicos esenciales. Los senderos más cortos tienen el menor número de conexiones posibles. A y B: Las conexiones se representa ponderadas o sin ponderación (de grosor variable) para significar la intensidad de la interacción biológica. C: Las conexiones pueden ser diraccionadas o no direccionadas. D: La relación entre más de un tipo de nodo (círculos y cuadros) pueden ser representadas en una red bipartita. EyF: Propiedades de redes aleatorias, la distribución P(k) sigue comportamiento bionoomial. La mayoría de las redes biológicas son sin escala con distribución P(k) por ley de poder. Solamente un número reducido de nodos (centros de actividead) están altamente conectados.
La tendencia al enracimamiento dentro de la red denota que exite conectividad reciproca en los racimos vecinos y establece una jerarquía. El coeficiente de enracimamiento local es usado para cuantificar la densidad es una red. Los patrones de interacciones nodales que se traducen en funciones biológicas específicas se denominan «motivos» y han demostrado ser útiles para predecir la función de una red, en especial las redes reguladoras. Las subunidades topológicas que están densamente conectadas se denominan «módulos» o «comunidades» (Ravasz, Somera, Mongru, & others, 2002). Representan un grupo de nódulos enlazados físicamente o funcionalmente que colaboran entre sí, para expresar un dado fenotipo funcional. Estos módulos, han hecho posible la identificaciń de funciones específicas como, coagulación, quimiotaxis, opsonización durante un análisis de redes, en la respuesta inflamatoria a infarto del miocardio (Azuaje, Rodius, Zhang, & others, 2011). Los análisis estadísiticos estándar, no pueden ser aplicados a redes que siguen una distribución gaussiana, pero sí la topología aleatoria de redes puede recavar significancia estadística de redes sin escala contra los de naturaleza aleatoria, cuando el tamaño de los sistema comparados son similares (Loscalzo & Barabasi, 2017).
Fenotipos Intermedios
El concepto de enfermedad cambia bajo los preceptos arriba señalados. Si bien toda enfermedad tiene un asidero genético y se recocen genes patogénicos dentro del interactoma humado, solamente un 10% de genes humanos se han podido asociar con una enfermedad conocida (<a href=»www.omin.org»>www.omim.org</a>) La mayoría de estos genes están ubicados periferícamente y no relacionados a centros de actividad (Goh, Cusick, Valle, Childs, Vidal, & Barabasi, 2007). La alta letalidad embrionaria de la disfunción en genes esenciales ha dificultado constatar la asociación de genes de nfermedad con centros de actividad, de manera especial en enfermedades crónicas que aparecen en edad avanzada. Analásis expansivos de matrices de expresión genética ha confirmado la presencia constante y omnipresente de «firmas de expresión genética» a lo largo de vasto y diferente tipo de tejido (Dudley, Tibshirani, Deshpande, & others, 2009). Según la «hipótesis de localidad», la tendencia de los genes patogénicos a interactuar con otros genes patogénicos de la red, conforma un «módulo funcional» el cual induce un patofenotipo que surge de la asociación Fig 3. De manera que la sola presencia de un gen(es) patogénico(s) en una red, no es equivalente que para un dado genotipo. se exprese un patofenotipo y se manisfieste enfermedad humana. Una gran sorpresa ha sido la constatación que algunos portadores de mutaciones Mendelianas consideradas de penetración completa y que se expresan en diferentes enfermedades de la infancia, poseen elasticidad y factores de protección que les permite llevar una vida normal. En un estudio de 589395 sujetos aparentemente normales, se identificaron 13 individuos con la firma genética para fibrosis quística, atelosteogénesis y síndrome de Smith-Lemli.Opitz., si bien, aún no se han podido identificar cuáles son estos factores de protección (Chen, Shi, Hakenberg, & others, 2016). En enfermedades complejas mediante GWAS (Genome-wide Association Studies) se ha identificado centenares de variantes asociadas con estas y se ha dilucidado su estructura genética. No obstante, la gran mayoría de las variantes expresa efectos de bajo riesgo y solamente una proporción de estas enfermedades se pueden clasificar como heredables. Para la mayoría de ellas, menos de un 10% de la varianza genética se explica por identificación de variantes comunes, quedando el grueso de heredabilidad, sin explicación. La reducida relación de probabilidades de 1.1. para héterozigotas y de 1.5-1.6 para homozigotas y las variantes latentes con probabilades casi nula de expresar enfermedad, explican la baja heredabilidad. Las variantes genéticas por sí mismas además, logran explicar sólo un 20% de la varianza general observada.
En teoría, una enfermedad rersultará de una o varias perturbaciones de un módulo de enfermedad. Pero la caracterización de un módulo disfuncional, a la hora de aplicar la teoría a la práctica, plantea retos formidables. Las representaciones estáticas o topológicas, ayudan en la introspección de los grandes datos derivados de las metodologías «ómicas» y mediante mapeo y herramientas de enracimamiento carentes de sego, se pueden identifcar sobreposiciones topológicas, las cuales pueden ser validadas contra módulos funcionales, ya previamente validadas con datos experimentales (Loscalzo, Kohane, & Barabasi, 2007) Fig C. La ubicación topológica de un gen determinado dentro del interactoma, puede tener un alto significado respecto a patogenia, eventual manifestación clínica de la enfermedad y las posibles interveciones terapéuticas, cuando además del gen disfuncional, setoma en cuenta las interacciones con otros módulos a los que se encuentra conectado. Pero es evidente de que si los trastornos Mendelianos clásicos se expresan en fenotipos variados como consecuencia de polimorfismo o mutaciones, tienen que intervenir otros factores en la conformación del fenotipo. Los factores ambiemtales asi como factores de comportamiento y factores cuantitavos de riesgo y las biopatologías genéricas que responden al estrés interno generado por los polimorfismo, mutaciones, agentes infecciosos, físicos-químicos y mecánicos, entre otros, deben de ser tomados en consideración.
Una visión global como trastorno de supersistema humano, faclita la compresión de la complejidad de la estructura que compone una enfermedad.
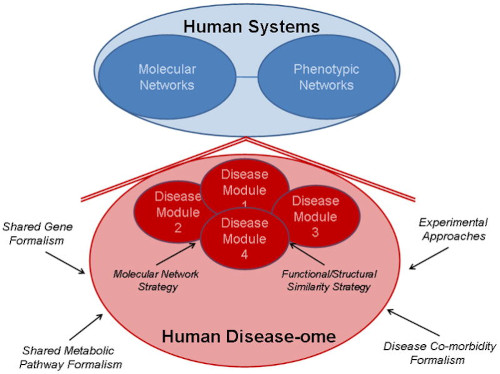
El Universo Biológico Humano de Sistemas
La redes moleculares y fenotípicas constituyen el Universo Humano de Sistemas Biológicos. Se relacionan entre ellos pero tienen su propia identidad. El total se compone de un conjunto de subredes. Los módulos disfuncionales pueden ser identificados por sus redes moleculares o su similitud funcional. Es necesario usar abordes basados sobre bio-informática para dar con los módulos. Estas son—el formalismo de genes compartdos, o el formalismode comorbilidades compartidas, o el formalismo de flujos metabólicos compartidos —o validando la bio-informática mediante experimentos de laboratorio.
La sensibilidad multifactorial es típica de los de los seres biológicos, de ahí su plasticidad a adaptarse y evolucionar según las condiciones en las cuales habitan. De la misma forma, el tejido acondicionado a responder multifactorialmente puede considerarse como terreno de nadie o encrucijada fértil, donde todo proceso biológico es posible; desde la desdiferenciación, el desarrollo, la resistencia, la elasticidad, la tolerancia, la resolución de la inflamación, la autoinmunidad y el cáncer. Es el trasfondo de la enfermedad, donde el interactoma se ve afectado en cuanto a la fluidez de la información a lo largo de la red y prioritariamente, require de la via resolutoria que devuelva la homeoestasis. La resolución puede ser adaptativa, de condiciones resultatntes mejoradas, pero que por el contrario la respuesta patobiológica puede desplazar la distribución de información, con retardo e incluso abolición de la adaptación/evolución y conducir a enfermedad crónica o muerte.Justo antes de que la enfermedad compleja exprese el fenotipo, su componente genético más las patobiologías genéricas que inciden sobre tal manifestación, abren ventanas de índole preventivo, o en el caso de poder intervenir en la resolución de las patobiologías, de intervenciones resolutorias que impactarán favorablemente sobre el fenotipo y que, muy probablemente, evitará la maladaptación. Esa zona gris, ha sido llamada plasticidad de fenotipos (Fox, Donelson, Schunter, Ravasi, & Gaitán-Espitia, 2019), mejor caracterizada por lo que se denomina como fenotipo interrmedio e ilustrado en la Fig (Loscalzo et al., 2007).
Dentro de la definición de fenotipo caben diferenciaciones que dependen de la verticalidad de la heredabilidad Mendeliana en manifestarse clínicamente, como cuando se trata de trastornos polimórficos que afectan a los procesos bioquímicos y moleculares, íntimamente dependientes de la mutación primaria. O en la mayoría de los casos, en que la mutación es sensible a procesos patobiológicos genéricos que responden al estrés organísmico generado y a las señales que se reciben por exposición ambiental. Estas respuestas a las patobiologías genéricas y que participan en la definición del fenotipo intermedio, son las respuestas a la inflamación, la trombosis, la fibrosis, la respuesta inmune (posresolución), la proliferación y la apoptosis/necrosis Fig. Comunmente entonces, una enfermedad es el resultado de influencias múltiples, de genotipos y de factores de riesgo cuantitativos de índole genérico, ambiental y hasta de comportamiento humano. Basarse en el fenotipo intermedio (toma en cuenta los factores de riesgo) para análisis estadístico, aumenta el poder estadístico de estudios de asociación entre genética y riesgo de que una enfermedad se manifieste (Li, Huang, & Amos, 2012).
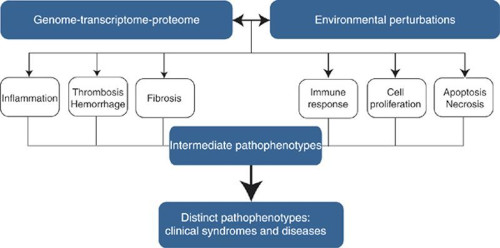
Diagrama que identifica las asociaciones entre factores genéricos y factores ambientales, que interactúan con los genotipos, los fenotipos intermedios y que colectivamente se manifiestan como patofenotipos definidos. Los fenotipos intermedios determinan, en grado variable, la variación en expresión de la manisfestación clínica, en individuos que poseen disfunción genética y que está sujeta a influencias del ambiente en el que habita (Loscalzo et al., 2007).
Información biológica
Si bien se ha descrito que el flujo de información en redes sin escala se facilita con tiempos mínimos de transición, hay que tomar en cuenta que las representaciones son prevalentemente estáticas, o es decir topológicas. Es indudable que este enfoque ha rendido mucha información valiosa y ha cambiado la forma de pensar. No obstante, en muchos aspectos se necesita ahondar en la dinámica y el flujo de información del interactoma, para obtener una mejor comprensión de la complejidad biológica y del concepto de enfermedad, mediante la simplificación que los sistemas de redes permiten. Es necesario conocer mejor los aspectos estructurales de las redes y deducir la interconectividad de la red, la potencia y la dirección de las interacciones nodales. Para muchas enfermedades se ha dilucidado el primer aspecto de esos atributos y parcialmente también del segundo. Pero ha sido hasta el momento, un reto formidable cuantificar el dinamismo cambiante de la señalización, en los sistemas de redes que simbolizan a las enfermedades humanas.
En cuanto al segundo punto hay que agregar que el conocimiento de la potencia de la señalización, se ha basado sobre extrapolaciones hechas en procariotas (bacterias) y eucariotas (levaduras) unicelulares, en los cuales las concentraciones de los metabolitos se miden con facilidad y precisión, que admiten cálculos de coeficientes de sensibilidad y los cuales predicen el efecto bioquímico causado por perturbaciones específicas de la red. Eso ha sido existoso en el contexto de patogénesis bateriológica y en especial a alteraciones globales metabólicas. que han esclarecido apsectos de crecimiento bacteriano, sensibilidad a antibióticos y la infectividad bacteriana en humanos. Deducir en tejido humano las redes metabólicas y recavar de ello conocimiento de patogenia, ha siso aún más arduo. Lo anterior es probablemente debido a que los metabolomas en muestras de tejido humano no han estado disponibles sino hasta muy recientemente, con el reparo de que inferencias acerca de las concentraciones iniciales de metabolitos, han resultado ser un tanto inciertas. Finalmente, debido a la complejidad de empatar senderos y aflujos sobreponiéndolos en tejido humano, que las constantes de velocidad son variables y están sujetas a alteracioens por actividad enzimática, a su expresión y a relaciones témporo-dependientes, la definición y la aplicación de flujo dinámico a redes genéticas reguladoras, han sido un desafío aún mayor.
El esclarecimiento del flujo de información es importante para completar el mapeo de los sistemas de redes. la patogenia de los fenotipos y el desarollo de interveciones reduccionistas. Pero cuando se toma en cuenta la ventana que se abre a nivel de fenotipos intermedios, ha salido a relucir información vital en cuanto a la dinámica del flujo de información y de cómo las intervenciones que modulen las patobiologías genéricas pueden prevenir las maladaptaciones. Pero antes de acuparnos de la dinámica del flujo de información, es necesario tomar en cuenta las sub-redes que caracterizan a las principales patobiologias: inflamación, trombosis y fibrosis.
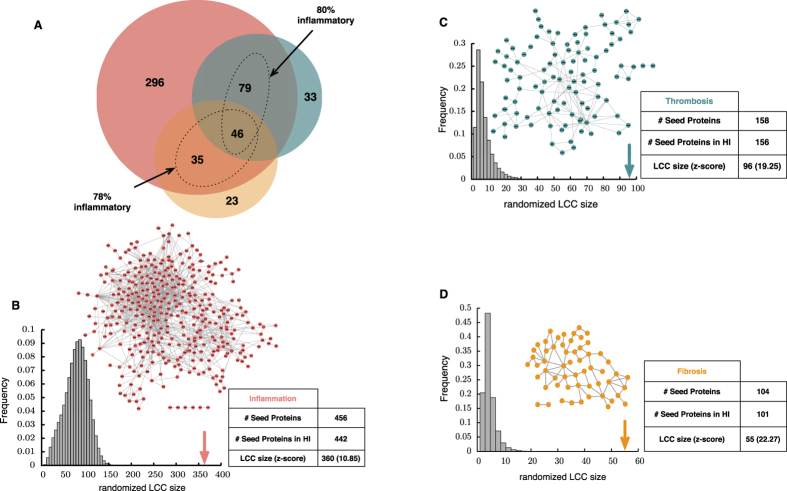
Ghiassan et al, con base en literatura altamente confiable y seleccionada, más la metodología DIAMOnD, compilaron las 3 subredes del interactoma humano reponsables de las respuestas patobiológicas de tipo inflamatorio, trombótico y fibroso. Además de constatar que se sobreponían densamente topológicamente, demostraron que son ricas de varios determinantes de enfermedad, los cuales incluyen: genes disfuncionales asociados con 50% de enfermedades complejas y genes diferenciados asociados a riesgo de enfermedad cardiovascular (i.e., enfermedad preclínica). Adicionalmente, genes incrustados relacionados con IL-6, IGF1, la activación extrínseca de la protrombina, la familia AP1 de factores de transcripción y el sendero PECAMI. Las proteínas asociadas al inflamasoma y fibrosoma, fueron identificadas como esenciales para mantener la estructura e integridad de la red. Al verificar la robustez de los módulos identificados mediante un método de árbol, encontraron que solamente las semillas inflamatorias son tronco-simil, mientras que las trombóticas y fibrosas son hoja-simil. Una proproción de 80% de las semillas fueron identificadas de tipo inflamatorio. Concluyeron mediante un análisis funcional de las abundantes proteínas asociadas con los módulos, que subyace omnipresente un mecanismo de índole inflamatorio en el meollo del fenotipo intermedio de la mayoría de las enfermedades complejas no transmisibles (Ghiassian, Menche, Chasman, & others, 2016).

Caracterización topológica de genes semilla en el contexto del Interactoma Humano.
(A) Diagrama Venn de genes semilla de tipo: infalamatorio(rojo), trombótioc(azul) y fibroso (naranja). (B–D) Corresponden a sub-gráficos de las 3 patobiologías. En conjunto forman un gran complejo interconectado sugerente de un vecindario altamente enriquecido de genes inflamatorios, trombóticos y fibróticos.
Información biológica y enfermedad
En vista que el estudio dinámico del flujo de información se encuentra en su infancia (Uda, 2020) y el enorme peso de la inflamación en los fenotipos inermedios, es conveniente recavar conclusiones, respecto de cómo la inflamación puede afectar la comunicación en el interactoma humano.

Señalización inflamatoria y homeostasis
(A) La señalización inflamatoria (IS) actúa en los mismo canales y nodos de la señalización hmeoestática (HS). A fiesn d ilustración el paralelo entre ambas es diagrama se refiere ala fuente de la IS como Contralador Inflamatorio (i.e. macrófago) en analogía al controlador hoeoestático (pancreas). (B) Los macrófagos producen TNF e IL.1 que ocupan los mismos flujos de la insulina, pero en dirección opuesta, lo que induce resistencia a la insulina y la supresión de almacenamiento de lípidos en el tejido adiposo mediante la inhibición de lipoproteina lipasa. TNF and IL-1 induce insulin resistance and suppress lipid storage in adipose tissue by inhibiting lipoprotein lipase. Al mismo tiempo sintonizan a las células pancreáticas β a reducir la cantidad de insulina que producen en relación al nivel de glucosa sanguíneo e intensificado por un menor aflujo de glucosa a la células β.
Los mediadores inflamatorios son antagónicos y dominantes sobre la señalización homestática. Los fines de estos procesos son discordantes. Ineludiblemente, la homeostasis debe de ser temporalmente desactivada ante una inflamación aguda de suficiente monta. La prioridad de eliminar retos infecciosos o trauma, antecede a la homeostasis. Por ejemplo, los cambios normales inducidos en la regulación de la temperatura corporal por el ambiente (termogénesis o sudor), son invalidados ante los cambios que la inflamación aguda puede inducir sobre el punto-set, al elevarlo y causar fiebre y termogénesis. La justificación de los ajustes que se observa ante un reto infeccioso, son presumiblemente debidos a la necsidad de variar la asignación de nutrientes; de tejidos de menor prioridad al aparato inmune de alta prioridad (Hotamisligil, Shargill, & Spiegelman, 1993). Es como una intesificación de lo que ya ocurre fisiológicamente, con el objetivo de reasignar nutrientes, como durante la gravidez, Por el contrario, la sensiilidad aumenta durante la restricción calórica y la pérdida de peso (Odegaard & Chawla, 2013; Watve & Yajnik, 2007).
En tejidos que desarrollan memoria inflamatoria (Sasson, Gordon, Christo, & others, 2020), las células recién reclutadas durante un reto sucesivo, pueden invertir los circuitos de comunicación del tejido con experiencia e inducir en este, un cambio de talante inmunitario, de innato a adaptativo. Las células CD8+ T residentes en el útero y la piel, dominan autónomamente durante un reto sucesivo, el reclutamiento de células, con preferencia de células T efectoras. La antigua predoninancia del receptor Toll-like/TLR), es revertida a predominancia del receptor de células T (TCR), lo que activa la inmunidad antiviral mediante células B, células dendríticas, células asesinas naturales y queratinocitos, junto con otras células residentes (Schenkel, Fraser, Beura, & others, 2014; Ariotti, Hogenbirk, Dijkgraaf, & others, 2014).
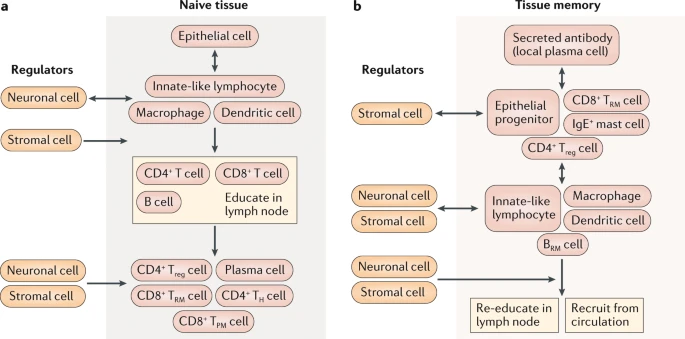 El lujo de información en tejido inocente (parte a) o el hipotético en tejido con experiencia (parte b), en el cual existe memoria inflamatoria con reguladores clave (enfatizados) del lado superior del flujo vertical. Las flechas indican la predominancia direccional del flujo de información. Mediante la memoria establecida en células plasma productoras de anticuerpos, las células residentes de memoria CD8+ T y las células progenitoras epiteliales, se revierte el flujo de información que existió en la parte a (tejido inocente). BRM cell, célula residente de memoria B; TH cell, células T ayudantes; TPM cell, células T periféricas de memoria; Treg cell, células T reguladoras; TRM cell, células T residentes de memoria (Ordovas-Montanes, Beyaz, Rakoff-Nahoum, & others, 2020).
El lujo de información en tejido inocente (parte a) o el hipotético en tejido con experiencia (parte b), en el cual existe memoria inflamatoria con reguladores clave (enfatizados) del lado superior del flujo vertical. Las flechas indican la predominancia direccional del flujo de información. Mediante la memoria establecida en células plasma productoras de anticuerpos, las células residentes de memoria CD8+ T y las células progenitoras epiteliales, se revierte el flujo de información que existió en la parte a (tejido inocente). BRM cell, célula residente de memoria B; TH cell, células T ayudantes; TPM cell, células T periféricas de memoria; Treg cell, células T reguladoras; TRM cell, células T residentes de memoria (Ordovas-Montanes, Beyaz, Rakoff-Nahoum, & others, 2020).
Evolución de la enfermedad cuando existe componente inflamatorio crónico
La evaluación de COPD en la cohorte ECLIPSE (Evalución de COPD Longitudinal para Identificar Desenlaces Predictivos NCT00292552) ha tenido una repecusión importante en dilucidar los subtipos de COPD (fenotipos), la estructuración genética e identificar nuevos marcadores de la enfermedad (Vestbo, Anderson, Coxson, & others, 2008). ECLIPSE fue el primer estudio observacional y longitudinal de COPD, en una población importante, el cual, después de la visita médica base, los participantes fueron eveluados posteriormente a los 3 meses, 6 mese y luego cada 6 meses por 3 años. Del esudio ECLIPSE, Agustí et al., determinaron en 1755 pacientes y como control en 297 fumadores y 202 no-fumadores, la prevalencia, la estabilidad temporal y el patrón de red (inflamasoma) con base en los 6 biomarcadores estudiados más frecuentemente en COPD: conteo de glóbulos blancos, CRP, IL-6. IL-8, fibrinógeno y TNFα, su asociación con las manifestaciones clínicas y la evolución final del paciente al cabo de 3 años. Los resultados fueron: 1) el inflamasoma de fumadores y pacientes con COPD son diferentes; 2) 30% de pacientes COPD no muestra inflamación, mientras que un 20% muestra presencia de inflamación persistente; y 3) a pesar de que existe estrecha similitud en las anormalidades estructurales y funcionales de ambos, los pacientes con marcadores de inflamación anormales, tuvieron una mortilidad por toda-causa 6 veces mayor a los que no presentaron signos de inflamación. Además existió el doble de exacerbaciones en el grupo de los pacientes con inflamación, durante los 3 años (Agustí, Edwards, Rennard, & others, 2012).
Dregan et al. utilizando la información de Biobank del Reino Unido, condujeron una investigación transversal para estimar el riesgo cardiometabólico y una cohorte para estimar el riesgo de mortalidad en pacientes con una afección inflamatoria crónica. En una población m=502341 un 4% (n=19, 082) aproximadamente presentó algún trastorno inflamatorio entre, soriasis (m=5286) la más común y SLE (n=654) lupus sistémico eritematoso. SLE mostró la asociación con riesgo múltiple (riesgo relativo (RR) 6.36, 95% CI 4.37 to 9.25) de trastorno cardiomet6abólico, seguido por RA artritis reumaoide (RR 1.70, 95% CI 1.59 to 1.83), UC colitis ulcerativa (RR 1.69, 95% CI 1.51 to 1.89), AS espondilitis anquilosante (RR 1.64, 95% CI 1.42–1.90) vasculitis(RR 1.64, 95% CI 1.42–1.90) y psoriasis (RR 1.25, 95% 1.16 to 1.35). La asociación fue más significativa entre quienes se les recetó AINES y corticoesteroides, SLE (RR 12.35, 95% CI 7.18 to 21.24), UC (RR 3.81, 95% CI 2.69 to 5.38), enfermedad de Crohn’s (RR 3.07, 95% CI 1.85 to 5.11), RA (RR 3.06, 95% CI 2.44 to 3.85), soriasis (RR 2.36, 95% CI 1.88 to 2.95), AS (RR 2.25, 95% CI 1.48 to 3.41) y vasculitis (RR 1.89, 95% CI 1.28 to 2.79). La mortalidad toda-causa fue mayor entre quienes presentaron efermedades inflamatorias crónicas (Dregan, Chowienczyk, & Molokhia, 2017).
En el estudio Moli-sani, una cohorte prospectiva basada en una población de 24325 mujeres y hombres de edad ≥35 años libre de inflamación aguda y de enfermedades hematológicas, desde marzo 2005 a abril 2010, se investigó la presencia de inflamación de bajo grado mediante la suma de 10 fichas de marcadores plasmáticos INFLA-score, CRP y recuentos de leucocitos y plaaquetas y la relación granulocitos/linfocitos. Valores anormales altos se consideraron como marcador de inflamación de bajo grado. Después del seguimineto por una media de 7.6 años, hubo 837 muertes por toda-causa. Los sujetos en la cuartilla alta de la calificaión de inflamación de bajo grado, mostraron un riesgo aumentado de la mortalidad en general (HR=1.44; 1.17–1.77), independientemente de confundidores. La magnitud del riesgo fue aún mayoor en individuos con biabetes2 (HR=2.48; 1.50–4.11) y enfermedad cardiovascular (HR=2.90; 1.74–4.84) en comparación con aquellos en la cuartilla más baja de la calificación. Es decir un aumento de riesgo 44% (23% a 83%) (Bonaccio, Di Castelnuovo, Pounis, & others, 2016).
Un estudio transversal en (n=631) hombres y mujeres y con seguimiento de 13 años, sobre disfunción endotelial e inflamación de bajo grado y la asociación con mortalidad cardiovascular, alcanzó un (relación de riesgo, 1.43 [95% CI, 1.17 to 1.77] por diferencia de 1 SD ) y en especial en diabéticos (Jager, Dekker, Kooy, Kostense, Nijpels, Heine, Bouter, & Stehouwer, 2006).
Farmacología revisitada
La carencia de nuevos fármacos que sean eficaces en enfermedades crónicas no transmisibles, obliga a apuntar a intervenciones que modulen la inflamación y logren resolver el trastorno evitando la posresolución y la entrada en escena de la inmunidad adaptativa. La posresolución es deseable en las barreras, pero cuando se trata de tejidos vitales de cuya vitalidad depende el correcto funcionamineto de un órgano, a toda costa se debe de evitar la fibrosis, patobiología genérica ampliamente ilustrada en esta entrega. El idioma metabólico parece ser apto para, mediante el potencial de membrana (ΔΨm) y el estado de energía ([ATP]/[ADP][Pi]) mitocondrial, en hablarle a tejido que se encuentra en un estado de aturdamiento comunicativo. La homeostasis se mantiene mediante un sistema sensorial/señalización bididerccional de control de red que llega a toda la organización metabólica celular. El estado de energía es mantenido con altísima precisión (a ∼1 parte en 1010) y responde a cambios transitorios de demanda (utlización de ATP) de más de 100 veces respecto del estado de reposo (Wilson, 2017).
Como arriba se expuso, en organismos con extensa diferenciación celular, el metabolismo sostiene, la robusta comunicación —célula a célula— necesaria para la estructuración y el funcionamiento integrado de tipo organísmico. Si bien el DNA constituye la plantilla para construir las partes, no logra responder con rapidez necesaria a las variaciones que provienen del ambiente. Las respuestas a este tipo de estímulo, deben ser inmediatas y en término de pocos segundos como también la respuesta adaptativa. El lenguaje metabólico bidireccional, es la única forma apta de comunicarse con tal rapidez y en tiempo real,en el microambiente tisular. Control en tiempo real, requiere de una unidad reguladora (Kotas & Medzhitov, 2015) con propiedades intrínsecas de establecer estabilidad mediante un punto-set y conectada al restante metabolismo de senderos metabólicos, que lo mantienen (Wilson, 2013).En eucariotas la única unidad con conexiones a todo el metabolismo celular es la oxidación fosforilativa, capaz de impulsar el estado de energía (extremo de larelación[ATP]/[ADP][Pi]) cerca de 105 M−1. Lo anterior equivale a 5 × 1010 mayor, dell valor de equilibrio: 2 × 10−6 M−1. En mamíferos, expansiones a más de 5 veces del punto-set resulta en disfuncionalidad y puede ser tolerado sólo por un tiempo muy limitado. De manera que, la oxidación fosforilativa puede impulsar el estado de energía hasta ([ATP]/[ADP][Pi]) un factor de 5 × 1010 por encima del equilibrio, pero al mismo tiempo mantiene el valor (media relativa al tiempo) dentro de un factor de 5. Se infiere de lo anterior que, la fosforilación oxidativa actúa como el mecanismo regulador metabólico en la célula y que el estado de energía es determinante, para la homeostasis celular (Wilson, 2015). Un modelo in vivo del mecanismo, ha demostrado ser altamente congruente con infomación experimental y también de la dependencia del mismo. respecto de la charca intramitocondrial de NAD, PO2 y el estado de energía. La alteración de la fosforilación oxidativa mediante carencia de substartos (oxigeno, NADH) o por disfunción enzimática y por la inflamación que causa el estrés metabólico, es conducente a trastornos patológicos de metabolismo celular y de funcionalidad tisular (Wilson, 2015). La fuerte dependencia de multitud de funciones, de su comunicación con el regulador del estado de energía, convierte a la señalización bidireccional, en óptimo instrumento de intervención en los microambientes que se hallan en estado de aturdamiento comunicativo y especialmente, si se acompaña de medidas estabilizadoras de la provisión de substrato, pH y control redox, congruentes con el microambiente.

Esquema miniαmizado de las funciones más importantes y la comunicación bi-direccional, señalización/control, del estado de energía. Todo metabolismo celular y tisular, está acoplado a y es regulado por el estado de energía. (Wilson, 2015)
Otra consideración, es la relativa a la condiciones de desbalance homeostático que los desajustes metabólicos causan y la inflamación que deriva de los mismos. Hoy se acepta que la inflamación puede derivar no solamente de causas externas tipo infección y trauma, pero también de factores internos, En el pasado se definía a la inflamación, en términos del estímulo, por lo que la definición era limitante e inadecuada y vinculada a la manifestación clásica de rubor. tumor, calor y dolor. El conocimiento de los mecanismos que median en la inflamación ha evolucionado, como también han cambiado los criterios para reconocer que existe inflamación; aún cuando los signos antes citados, están ausentes. Se ha reportado la existencia de inflamación de bajo grado, en una gran variedad de circunstancias. Entre muchas otras, la lista comprende, fumado activo y pasivo, deprivación de sueño, poca actividad física, fibrilación auricular, hipertensión, bajo peso al nacimiento, hernia de disco lumbar, ovarios poliquísticos, residencia en altitud alta, apnea obstructiva del sueño, síntomas pre-menstruales, una variedad de dietas poco saludables, hipoxia, cambios de osmolaridad, exceso de sal, alcohol, estrés emocional, aislameinto social y edad avanzada (Antonelli & Kushner, 2017). Toda desviación de la homeostasis es reconocida por una serie de células sentinelas que comprenden, macrófagos, células dendríticas, células linfoides innatas ILC y las cuales entran en un estado de estrés, tendiente a reestablecer la homeostasis. El estrés es advertido de manera especial en el retículo endoplásmico (ER), donde el sistema de señalización de proteína no-plegada (UPR) emite señales multinivel mediante la inducción de genes y citocinas asociados a la puesta en marcha de la respuesta de fase aguda. El factor NF-kB, reguladorr transcripcional maestro de la inflamación, puede ser activado por los 3 senderos UPR. El estrés endoplásmico puede activar la familia de receptores nod-simil, dominio que contiene 3 complejos (NLRP3) (Wang & Kaufman, 2016). El elemento de respuesta a cAMP proteína ligante-H (CREBH), factor transcripcional similar a ATF-6 (factor transcriñcional cAMP dependiente ATF-6α); uno de 3 sensores alojados en la membrana del ER que gatilla la UPR, actúa mediante respuesta de fase aguda hepática y la transcripcción de las proteinas CRP y hepcidina (Shin, Chung, Joe, & others, 2012). El factor CREBH responde al estrés metabólico de lípidos mediante la expresión de genes involucrados en la lipogénesis, oxidación de ácidos grasos y lipolisis a nivel hepático (Zhang, Wang, Zheng, & others, 2012). El tejido adiposo en obesos se caracteriza por la expresión aumentada del TNF y la IL-6 y otros mediadores inflamatorios (Gregor & Hotamisligil, 2007). Finalmente, el calcio liberado del ER aumeta la producción de especies reactivas oxidativas (Zhang & Kaufman, 2008). Entre los estresores metabólicos que causan estrés en el ER, se menciona: deprivación de glucosa, perturbaciones del nivel de calcio intraluminal, citocinas, estado celular redox alterado, hipoxia, toxinas, virus, aumento del tráfico proteíco y exceso o deficiencia de nutrientes (Hotamisligil, 2006).
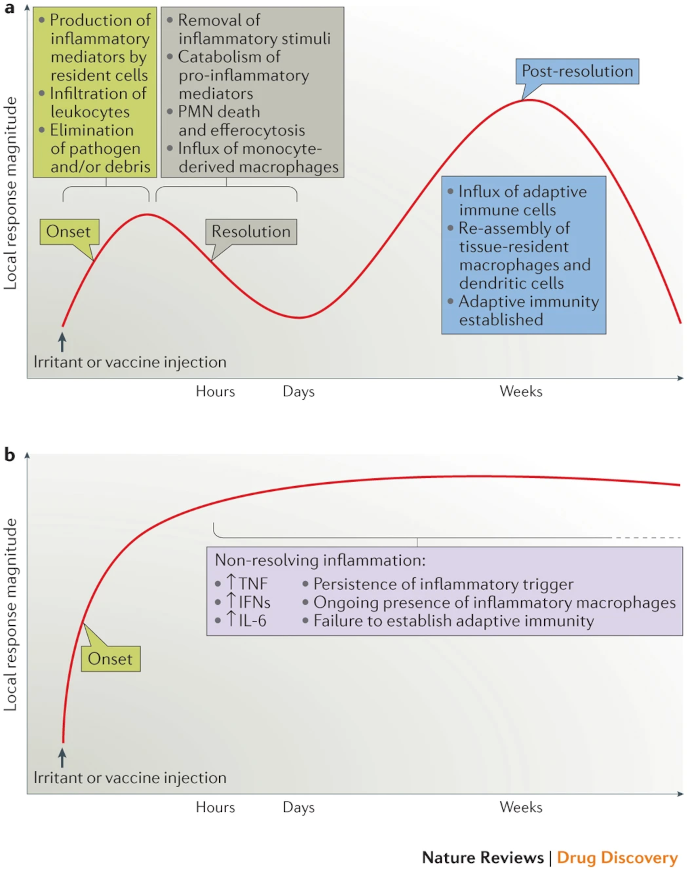
 También contrariamente a lo que se ha creído por siglos, la resolución de la inflamación no siempre conduce a las condiciones pre-existentes de homeostasis.Según los evolucionistas, el ser humano no fue diseñado a imagen un modelo de perfección, pues es el resultado de cambios sobre modelos pre-existentes que sucesivamente las mutaciones, el flujo genético, la variación genética y la recombinación condujo al humano moderno, en lapso de varios millones de años. O según François Jacob, es el resultado de la adaptación de secuencias pre-existentes, o como lo expresó: «La naturaleza es un tequio, pero no un inventor». Salta a la mente, que de ser exacto lo propuesto, en un organismo desarrollado en esa manera, no siempre los desenlaces van a ser perfectos. Muchos mecanismos de control, ciclos de retroalimentación, inhibidores moleculares, el UPR, proteinas de golpe de calor, necesitan ser armonizad@s a nivel celular, de tejido y de organismo. Por muchísimo tiempo, se supo cómo se induce la inflamación y creímos firmemente, que se resolvía pasivamente. En las últimas 3 décadas, aprendimos que la resolución es un proceso activo con mediadares lipídicos específicos que inducen un desenlace (Serhan & Levy, 2018). Durante la última, aprendimos que la inflamación no termina con la fase inicial mediada por la inmunidad innata, pero que existe una fase pos-resolución, que deja una ventana abierta para que la inmunidad adaptativa haga su aporte, cuando este es requerido (Newson, Motwani, Kendall, Nicolaou, Muccioli, Alhouayek, Bennett, Van De Merwe, James, De Maeyer, & Gilroy, 2017; Newson, Stables, Karra, & others, 2014). Viendo el gráfico Fig, parece que en la figura, pareciera un espejo ontológico de la evolución de la inmunidad; la innata muy conservada y antígua, la adaptativa mucho más reciente e insertada a la respuesta, prostrimeramente (Fullerton & Gilroy, 2016).
También contrariamente a lo que se ha creído por siglos, la resolución de la inflamación no siempre conduce a las condiciones pre-existentes de homeostasis.Según los evolucionistas, el ser humano no fue diseñado a imagen un modelo de perfección, pues es el resultado de cambios sobre modelos pre-existentes que sucesivamente las mutaciones, el flujo genético, la variación genética y la recombinación condujo al humano moderno, en lapso de varios millones de años. O según François Jacob, es el resultado de la adaptación de secuencias pre-existentes, o como lo expresó: «La naturaleza es un tequio, pero no un inventor». Salta a la mente, que de ser exacto lo propuesto, en un organismo desarrollado en esa manera, no siempre los desenlaces van a ser perfectos. Muchos mecanismos de control, ciclos de retroalimentación, inhibidores moleculares, el UPR, proteinas de golpe de calor, necesitan ser armonizad@s a nivel celular, de tejido y de organismo. Por muchísimo tiempo, se supo cómo se induce la inflamación y creímos firmemente, que se resolvía pasivamente. En las últimas 3 décadas, aprendimos que la resolución es un proceso activo con mediadares lipídicos específicos que inducen un desenlace (Serhan & Levy, 2018). Durante la última, aprendimos que la inflamación no termina con la fase inicial mediada por la inmunidad innata, pero que existe una fase pos-resolución, que deja una ventana abierta para que la inmunidad adaptativa haga su aporte, cuando este es requerido (Newson, Motwani, Kendall, Nicolaou, Muccioli, Alhouayek, Bennett, Van De Merwe, James, De Maeyer, & Gilroy, 2017; Newson, Stables, Karra, & others, 2014). Viendo el gráfico Fig, parece que en la figura, pareciera un espejo ontológico de la evolución de la inmunidad; la innata muy conservada y antígua, la adaptativa mucho más reciente e insertada a la respuesta, prostrimeramente (Fullerton & Gilroy, 2016).
Los experimentos en ratones de Newson et al. sobre una tercera fase de aflujo de células adaptativas en inflamación provocada por inyección peritoneal de zimozan, son muy elocuentes. En primer lugar es de considerar que la respuesta es dependiente de la dosis, y que la pos-resolución se establece con dosis bajas y ausencia de la ventana adaptativa a dosis altas (x100). En la última el perfil de tipo M1-simil Mφs y la secreción de altos niveles de TNF-α, IFN-γ, e IL-6 (>2000 pg/mL) y el número reducido de Tregs y linfocitos efectores/memoria, sugieren fuertemente de un estado de inmunosupresión. Al respecto es ampliamente conocido que el perfil claramente proinflamatorio de las citocinas, como el IFN y TNF-α. son antagonistas a la activación de respuestas de las células T (González-Navajas, Lee, David, & others, 2021). Según luce, la inmunidad innata resuelve completamente la inflamación aguda sin necesidad de recurrir a la ventana adaptativa, la cual es inclusive reprimida para evitar el peligro de autoinmunidad. Ver figura abajo(Feehan & Gilroy 2019).
.
En el caso de la dosis menor, existió un aflujo de moMφs derivados de Ly6ch+, moDCs, y MDSCs del sexto día en adelante. Adicionalmente los resMφs (de derivación pre-natal) reaparecen, luego de haberse desvanecido durante la respuesta inflamatoria. Junto con el aflujo adaptativo se observó expansión linfonodal y un aumento de celulas de memoria periféricas y linfocitos reguladores. Junto con la expansión linfonodal los autores observaron la aparición de «manchas de leche» en el omento. Lo anterior se acompaña a una migración de monocitos que expresan la quimiocina CCR2. Poco despuŕs los resMφs y los moMφs expresan la FoxP3 en las células CD4 T, mientras los moDcs gatillan su proliferación. Los resMφs se expanden a toda la cavidad peritoneal y una porción de estos, migran a los linfonodos e inician la contracción linfonodal, en una modalidad iNOS-dependiente, del día 9 al 13 en adelante (Newson et al., 2014). Lo anterior acompañado a activación aumentada de la E sinatasa-1 en los microsomas (mPFES-1 y -2) junto con aumento de secreción de ciclooxigenasa COX.1 en los macrófagos, lo cual resulta en biosíntesis sostenida de prostaglandina (PG)E2. La elevación de prostanoides en la fase de posresolución, está dominada por la COX-1 y las isformas mPFES-1 y -2. Paralelo a la infiltración de monocitos existió, a partir del día 3, infiltación de células asesinas naturales (NK) y células T CD4 y CD(8, contendencia a disminuir a partir del día (Newson et al., 2017).
En humanos, la inflamación aguda que sigue a la inyección i.d. de E. coli muerto por UV, en el antebrazo de voluntarios sanos y que suscita todos los cambios clásicos de la inflamación aguda y la infiltración de PMNs y citocinas/quimiocinas con un pico a las 4 hrs, aparentemente se resuelve en un término de 24-72 hrs. No obstante, días después puede ser observada en el sitio de inyección (Sasson et al., 2020) la infiltración de macrófagos y escasos PMNs y elevación de los prostanoides, resolvinas D (RvD5) y lipoxinas B (LXB4) (Motwani, Newson, Kwong, Richard-Loendt, Colas, Dalli, & Gilroy, 2017). En comparación al tejido virgen, los signos inflamatorios y la vesícula, se ven marcademente disminuidos y existe una respuesta adaptada al nuevo reto en un tejido que que posee «memoria del tejido», caraterístico de la memoria inflamatoria. Al enfrentar el nuevo reto, el tejido responde de acuerdo a la información que guardó del encuentro previo, pudiendo variar la naturaleza de la respuesta según el caso específico y las condiciones del microambiente y la cual puede ser: adaptativa, tolerante o maladaptativa (Casey, Fraser, Schenkel, Moran, Abt, Beura, Lucas, Artis, Wherry, Hogquist, Vezys, & Masopust, 2012), (Ariotti et al., 2014), (Souquette & Thomas, 2018), (Sasson et al., 2020)
.Es muy elocuente el hallazgo de las manchas de leche MS en el omento de los ratones inyectados intraperitonealmente con zimozan. Son estructuras linfoides inducidas, como las iBALT (Hwang, Randall, & Silva-Sanchez, 2016), (Silva-Sanchez & Randall, 2020), que en este ámbito, destaca la tendencia de que se establezca protección adaptativa, dondequiera exista un estímulo generado por agentes externos (Rangel-Moreno, Moyron-Quiroz, Carragher, & others, 2009). Dependiendo del tejido linfoide, hay varíantes de cómo se captan antígenos en el aparato adaptativo; mediante linfáticos aferentes en los linfonodos. Los tejidos linfoides de las mucosas, lo hacen a través de epitelio especializado y las MS, captan directamente los antígenos que puedan estar suspendidos en el fluido peritoneal. En el caso de células activadas por LPS, la migración a las MS, puede ser dirgida por receptores de quimiocinas, o pueden simplemente captarlas por arrastre del flujo peritoneal (Rangel-Moreno et al., 2009). Es notable la aptitud de las mucosas, en desarrollar sea inmunidad de tipo innato que adaptativo y la poca atención que se le presta a dicha oportuniad en la práctica para inducir protección, y en manera especial, a retos respiratorios altos. Aplicando los principios que se expondrán, es perfectamente posible sacar partido de la proximidad que existe entre agentes externos y las estructuras linfoides en la mucosa naso/orofaríngea. Mediante el uso de formulaciones mucoadherentes, pueden ser presentados al sistema adaptativo durante el transcurso de una reto natural en el aparato alto. Según la experiencia desarrollada en BioAcyl Corp, sin la necesidad de recurrir a medidas inmunogénicas, es posible mediante dichas formulaciones, inducir protección robusta y duradera contra los retos altos, extensiva al aparato respiratorio bajo.
Si en la mucosa se aplican medidas de contol del microambiente junto a la señalización metabólica, es perfectamente factible montar una intervención a nivel de fenotipo intermedio que mediante señales de pocos segundos, resuelva el problema comunicativo en un tejido con el interactoma en estado de aturdimiento y que logre la señalización oportuna que conduzca a la resolución de la inflamación. Adicionalmente el encuento con un reto natural (en el caso de la mucosa del aparato alto), se establece memoria de tejido que otorga protección de amplio espectro, robusta y duradera a retos sucesivos, tanto a cargo del sistema innato que del adaptativo. La encrucijada adaptativa que se describe en el párrafo anterior, es típica de tejido con el interactoma afectado en su poder de comunicación, ya que el desenlace en un nteractoma perturbado puede tomar cualquiera de los giros maladaptativos, de los cuales sobran ejemplos descritos en la literatura científica. Con la pérdida de homeostasis, vence el que se adueñe del control metabólico y la señalización; como sucede con el cáncer. Al considerar lo vital, como lo es el estado de energía de la célula así como el microambiente que se necesita para mantener la homeostasis, las intervenciones que pueden vencer esas profundas alteraciones de comunicación más el estrés que autoinician las patobiologías genéricas, deben de estar dirigidas a restablecer la señalización apta que enrumbe a la resolución completa de la inflamación y a corregir las condiciones perturbadas del microambiente; principalmente laprovisión de substrato, pH y control redox.
Congruente con el derrotero que los fenotipos pueden tomar cuando no hay estabilidad en el interactoma y el microambiente se encuentra alterado, es el hallazgo de 4 tipos de perfil para las células derivadas de monocitos e indentificados en la fase de la posresolución, del estudio de Newson et al. Los resMφs, que expresan TGF-α e IL-10. En contraste, los moMφs que expresan IL12p35, Las moDCs, que expresan IL1α, CCR7, CCR2, y CCL7. Los moMφs, que persistieron en la cavidad por meses, demostraron un fenotipo diferente a los resMφs y moMφs, el cual tampoco coincidían con el perfil de M1 y M2. Los autores concluyeron, en que la diversidad de fenotipo en las células de origen monocitario, puede relacionarse más con el estadío de inflamación en que se encuentra el trastorno y con el nicho que ocupe el macrófago en el tejido, más las condiciones específicas del microambiente (Okabe & Medzhitov, 2014)
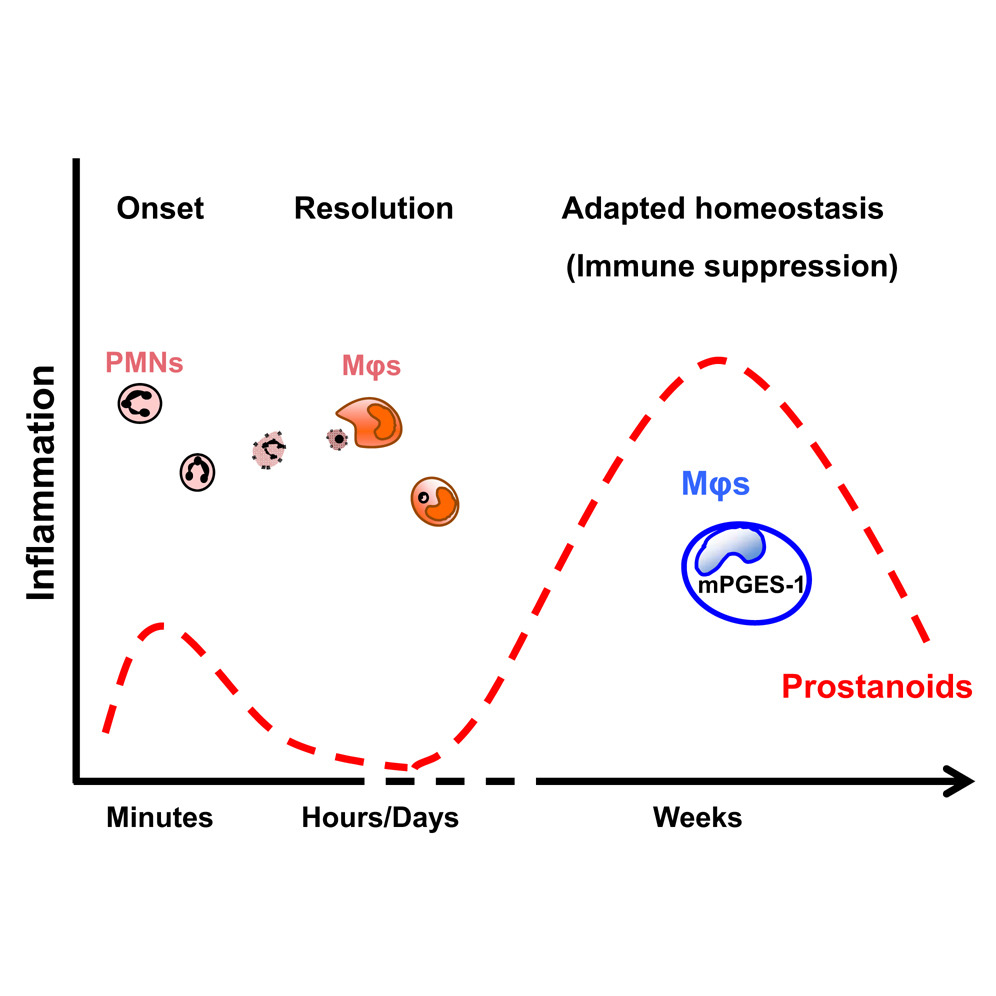
Fig que repretesenta en forma simplificada la relación entre macrófagos y la fase posresolución.
De acuerdo a este enfoque actualizado del concepto de enfermedad, el concepto reduccionista de ligando/receptor, o de «bala de plata» o Zauberkugel postulado en 1909 por Paul Erlich (Premio Nobel y pionero de la química medicinal), a pesar de haber guiado al desarrolo de fármacos por más de un siglo, flaquea ante el nuevo conocimiento y los nuevos retos. Agentes mono-especificos para tratar enfermedades multifactoriales, es un concepto que comienza a ser superado y cuya utilidad necesita ser redimesionada (Leopold & Loscalzo, 2018).
Hacia intervenciones homeoadaptativas
Una intervención homeoadaptativa es la que evita la maladaptación en primer lugar y luego de controlar la inflamación (ubicua en homeostasis alterada (Chaudhari, Talwar, Parimisetty, & others, 2014), devuelve al organismo a una situación de homeostasis mejorada con aptitud de enfrentar mejor, retos sucesivos. Debe de ser una intervención priva de efectos secundarios o que afecte negativamente otras funciones del organismo. Puede ser tratamiento único o, idealmente. combinado a autacoides.
BioAcyl Corp, ha desarrollado mediante control de la inflamación con base en señalización metabólica y el control de microambiente en los parámetros de pH y eH, opciones puestas en la práctica común en humanos, para cicatrización, lesiones músculoesqueléticas, manejo de dolor neuropático, afeccciones respiratorias altas con protección pumonar, manejo de salud buco-dental, manejo de acné, manejo de vaginosis, acondicionamiento de uñas, problemas dermatológicos, problemas hemorroidales, formulaciones especialmente diseñadas para el área involucrada. Con las formulaciones usadas según nuestras recomendaciones, los resultados se obtienen sin uso de antibióticos, AINES, corticoesteriodes y opiaceos. Existen combinaciones con otros autacoides que tienen características muy parecidas a los elementos que se emplean en las formulaciones, enteramente libres de fectos no deseados.
Por ser intervenciones dirigidas a los fenotipos intermedios y las patobiologías genéricas, la respuesta es universal y muy poca es la variación individual. Son intervenciones que pueden ser usadas indeterminadamente sin interrupción por largo tiempo, o en el cuidado de mucosas, de forma amigable y por toda una vida.
Células NIH 3T3 de ratón (fibroblastos), fueron heridas utilizando una hoja de afeitar y para herir el manto de las células, marcar el sitio de la herida y permitir que las células próximas al área herida migren al espacio vacío. Para crear la capa confluente, las células se sembraron en placas de cultivo pre-recubiertas con 10μ/ml de AcP2 disuelto en agua y se cultivaron durante 24 horas y las células dejaron de dividirse por inhibición de contacto celular. Después de 24 horas, se incubaron adicionalmente en medio de cultivo celular que carecía de suero de ternera fetal y se dejaron sin suero durante otras 20 horas. Para el modelo de control, otras células se sembraron en placas prerrecubiertas con medio de cultivo celular simple e igualmente en condiciones sin suero. Se repitió el procedimiento e igual las heridas anteriores, pero sin presencia de AcP2. Se descubrió que la migración de fibroblastos al área de la herida simulada en platos pre-recubiertos con AcP2 era más significativa que en los controles. Más células cultivadas en platos pre-recubiertos con AcP2 (indicado como MH) migraron a una distancia más larga a través del borde20 h despuésde la herida, que las células de control (indicado como VE). El número de células que migraron a través del borde de la herida se cuantificó en incrementos de 0,1 mm distal al borde de la herida.

Es alentador haber redescubierto aspectos nuevos para fármacos que en esencia pueden ser considerados como típicos ligando/receptor y abren posibilidades renovadoras. Queda por constatar, si los beneficios que se han observado tienen el alcance de asegurar el desenlace que procure la homeo-adaptación; es decir, resolución sin visos de maladaptación. Para ilustrar que significa lo anterior y para concluir, el video que se presenta es paradigmático de lo que significa pérdida de homeostasis y recuperación de homeostasis adaptada. Los varios ejemplos hablan por si mismos y colocan a los productos de BioAcyl Corp en una categoría avanzada de intervenciones a nivel de fenotipo intermedio.
Agustí, A., Edwards, L. D., Rennard, S. I., & others. (2012). Persistent Systemic Inflammation is Associated with Poor Clinical Outcomes in COPD: A Novel Phenotype. PloS one, 7(5), e37483.
Albert, R., Jeong, H., & Barabasi, A.-L. (2000). Error and attack tolerance of complex networks. Nature, 406(6794), 378—382.
Albert, R., & Barabasi, A.-L. (2002). Statistical mechanics of complex networks. Rev. Mod. Phys. 74(1), 47—97.
Antonelli, M., & Kushner, I. (2017). It’s time to redefine inflammation. FASEB J. 31(5), 1787—1791.
Ariotti, S., Hogenbirk, M. A., Dijkgraaf, F. E., & others. (2014). Skin-resident memory CD8+ T cells trigger a state of tissue-wide pathogen alert. Science, 346(6205), 101—105.
Ataullakhanov, F. I., & Vitvitsky, V. M. (2002). What Determines the Intracellular ATP Concentration. Bioscience reports, 22(5-6), 501—511.
Atkinson, D. E., & Walton, G. M. (1967). Adenosine Triphosphate Conservation in Metabolic Regulation RAT LIVER CITRATE CLEAVAGE ENZYME. Journal of Biological Chemistry, 242(13), 3239—3241.
Azuaje, F. J., Rodius, S., Zhang, L., & others. (2011). Information encoded in a network of inflammation proteins predicts clinical outcome after myocardial infarction. BMC Med. Genomics, 4, 59.
Barabasi, A.-L., & Albert, R. (1999). Emergence of Scaling in Random Networks. Science, 286(5439), 509—512.
Barabasi, A.-L., Gulbahce, N., & Loscalzo, J. (2011). Network Medicine: A Network-based Approach to Human Disease. Nat. Rev. Genet. 12(1), 56.
Becker, A., Thakur, B. K., Weiss, J. M., Kim, H. S., Peinado, H., & Lyden, D. (2016). Extracellular Vesicles in Cancer: Cell-to-Cell Mediators of Metastasis. Cancer Cell, 30(6), 836—848.
Bonaccio, M., Di Castelnuovo, A., Pounis, G., & others. (2016). A score of low-grade inflammation and risk of mortality: prospective findings from the Moli-sani study. Haematologica, 101(11), 1434.
Casey, K. A., Fraser, K. A., Schenkel, J. M., Moran, A., Abt, M. C., & Beura, L. K., et al. (2012). Antigen independent differentiation and maintenance of effector-like resident memory T cells in tissues. Journal of immunology (Baltimore, Md. : 1950), 188(10), 4866.
Chaudhari, N., Talwar, P., Parimisetty, A., & others. (2014). A Molecular Web: Endoplasmic Reticulum Stress, Inflammation, and Oxidative Stress. Front. Cell. Neurosci. 8,
Chen, R., Shi, L., Hakenberg, J., & others. (2016). Analysis of 589,306 genomes identifies individuals resilient to severe Mendelian childhood diseases. Nat. Biotechnol. 34(5), 531—538.
De la Fuente, I. M. (2015). Elements of the cellular metabolic structure. Frontiers in Molecular Biosciences, 2,
DeBerardinis, R. J., & Thompson, C. B. (2012). Cellular metabolism and disease: what do metabolic outliers teach us? Cell, 148(6), 1132.
Dregan, A., Chowienczyk, P., & Molokhia, M. (2017). Cardiovascular and type 2 diabetes morbidity and all-cause mortality among diverse chronic inflammatory disorders. Heart, 103(23), 1867—1873.
Dudley, J. T., Tibshirani, R., Deshpande, T., & others. (2009). Disease signatures are robust across tissues and experiments. Mol. Syst. Biol. 5(1), 307.
Feehan, K. T., & Gilroy, D. W. (2019). Is resolution the end of inflammation. Trends in Molecular Medicine, 25(3), 198–214.
Fox, R. J., Donelson, J. M., Schunter, C., Ravasi, T., & Gaitán-Espitia, J. D. (2019). Beyond buying time: the role of plasticity in phenotypic adaptation to rapid environmental change. Phil. Trans. R. Soc. B, 374(1768).
Fullerton, J. N., & Gilroy, D. W. (2016). Resolution of inflammation: a new therapeutic frontier. Nat. Rev. Drug Discovery, 15(8), 551—567.
Ghiassian, S. D., Menche, J., Chasman, D. I., & others. (2016). Endophenotype Network Models: Common Core of Complex Diseases. Sci. Rep. 6,
Gibbons, J. J., Abraham, R. T., & Yu, K. (2009). Mammalian Target of Rapamycin: Discovery of Rapamycin Reveals a Signaling Pathway Important for Normal and Cancer Cell Growth. Semin. Oncol. 36, S3—S17.
Goh, K.-I., Cusick, M. E., Valle, D., Childs, B., Vidal, M., & Barabasi, A.-L. (2007). The human disease network. Proc. Natl. Acad. Sci. U.S.A. 104(21), 8685—8690.
González-Navajas, J., Lee, J., David, M., & others. (2021). Immunomodulatory functions of type I interferons. Nat. Rev. Immunol. 12(2), 125.
Gregor, M. F., & Hotamisligil, G. S. (2007). Thematic review series: Adipocyte Biology. Adipocyte stress: the endoplasmic reticulum and metabolic disease. J. Lipid Res. 48(9), 1905—1914.
Hotamisligil, G. S., Shargill, N. S., & Spiegelman, B. M. (1993). Adipose expression of tumor necrosis factor-alpha: direct role in obesity-linked insulin resistance. Science, 259(5091), 87—91.
Hotamisligil, G. S. (2006). Inflammation and metabolic disorders. Nature, 444(7121), 860—867.
Hwang, J. Y., Randall, T. D., & Silva-Sanchez, A. (2016). Inducible Bronchus-Associated Lymphoid Tissue: Taming Inflammation in the Lung. Frontiers in immunology, 7,
Jacob, F. (1977). Evolution and tinkering. Science, 196(4295), 1161—1166.
Jager, J. D., Dekker, J. M., Kooy, A., Kostense, P. J., Nijpels, G., & Heine, R. J., et al. (2006). Endothelial dysfunction and low-grade inflammation explain much of the excess cardiovascular mortality in individuals with type 2 diabetes. Arteriosclerosis, Thrombosis, and Vascular Biology, 26(5), 1086—1093.
Jones, D. P., & Sies, H. (2015). The Redox Code. Antioxidants & Redox Signaling, 23(9), 734—746.
Kotas, Maya E., & Medzhitov, R. (2015). Homeostasis, Inflammation, and Disease Susceptibility. Cell, 160(5), 816—827.
Lane, N., & Martin, W. F. (2012). The Origin of Membrane Bioenergetics. Cell, 151(7), 1406—1416.
Li, Y., Huang, J., & Amos, C. I. (2012). Genetic Association Analysis of Complex Diseases Incorporating Intermediate Phenotype Information. PloS one, 7(10), e46612.
Liu, H., Du, Y., St-Pierre, J. .-P., Bergholt, M. S., Autefage, H., & Wang, J., et al. (2020). Bioenergetic-active materials enhance tissue regeneration by modulating cellular metabolic state. Science Advances, 6(13), eaay7608.
Loscalzo, J., & Barabasi, A.-L. (2017). Network medicine. Harvard University Press.
Loscalzo, J., Kohane, I., & Barabasi, A.-L. (2007). Human disease classification in the postgenomic era: A complex systems approach to human pathobiology. Mol. Syst. Biol. 3(1), 124.
Loscalzo, J., & Barabasi, A.-L. (2011). Systems Biology and the Future of Medicine. Wiley Interdiscip. Rev. Syst. Biol. Med. 3(6), 619.
Lum, J. J., Bauer, D. E., Kong, M., Harris, M. H., Li, C., & Lindsten, T., et al. (2005). Growth Factor Regulation of Autophagy and Cell Survival in the Absence of Apoptosis. Cell, 120(2), 237—248.
Lyssiotis, C. A., & Kimmelman, A. C. (2017). Metabolic Interactions in the Tumor Microenvironment. Trends Cell Biol. 27(11), 863—875.
Martin, W., & Müller, M. (1998). The hydrogen hypothesis for the first eukaryote. Nature, 392(6671), 37—41.
Motwani, M. P., Newson, J., Kwong, S., Richard-Loendt, A., Colas, R., & Dalli, J., et al. (2017). Prolonged immune alteration following resolution of acute inflammation in humans. PloS one, 12(10), e0186964.
Newson, J., Stables, M., Karra, E., & others. (2014). Resolution of acute inflammation bridges the gap between innate and adaptive immunity. Blood, 124(11), 1748—1764.
Newson, J., Motwani, M. P., Kendall, A. C., Nicolaou, A., Muccioli, G. G., & Alhouayek, M., et al. (2017). Inflammatory Resolution Triggers a Prolonged Phase of Immune Suppression through COX-1/mPGES-1-Derived Prostaglandin E2. Cell Rep. 20(13), 3162—3175.
Nikoletopoulou, V., Markaki, M., Palikaras, K., & Tavernarakis, N. (2013). Crosstalk between apoptosis, necrosis and autophagy. Biochimica et Biophysica Acta (BBA) – Molecular Cell Research, 1833(12), 3448—3459.
Odegaard, J. I., & Chawla, A. (2013). Pleiotropic Actions of Insulin Resistance and Inflammation in Metabolic Homeostasis. Science, 339(6116), 172—177.
Oikonomou, P., & Cluzel, P. (2006). Effects of topology on network evolution. Nat. Phys. 2, 532—536.
Okabe, Y., & Medzhitov, R. (2014). Tissue-Specific Signals Control Reversible Program of Localization and Functional Polarization of Macrophages. Cell, 157(4), 832—844.
Ordovas-Montanes, J., Beyaz, S., Rakoff-Nahoum, S., & others. (2020). Distribution and storage of inflammatory memory in barrier tissues. Nat. Rev. Immunol. 20(5), 308.
Özalp, V. C., Pedersen, T. R., Nielsen, L. J., & Olsen, L. F. (2010). Time-resolved Measurements of Intracellular ATP in the Yeast Saccharomyces cerevisiae using a New Type of Nanobiosensor. Journal of Biological Chemistry, 285(48), 37579—37588.
Pastor-Satorras, R., & Vespignani, A. (2002). Epidemic dynamics in finite size scale-free networks. Phys. Rev. E, 65(3), 35108.
Rangel-Moreno, J., Moyron-Quiroz, J. E., Carragher, D. M., & others. (2009). Omental Milky Spots Develop in the Absence of Lymphoid Tissue-Inducer Cells and Support B and T Cell Responses to Peritoneal Antigens. Immunity, 30(5), 731—743.
Ravasz, E., Somera, A. L., Mongru, D. A., & others. (2002). Hierarchical Organization of Modularity in Metabolic Networks. Science, 297(5586), 1551—1555.
Sasson, S. C., Gordon, C. L., Christo, S. N., & others. (2020). Local heroes or villains: tissue-resident memory T cells in human health and disease. Cell. Mol. Immunol. 17(2), 113—122.
Schenkel, J. M., Fraser, K. A., Beura, L. K., & others. (2014). Resident memory CD8 T cells trigger protective innate and adaptive immune responses. Science, 346(6205), 98—101.
Serhan, C. N., & Levy, B. D. (2018). Resolvins in inflammation: emergence of the pro-resolving superfamily of mediators. J. Clin. Invest. 128(7), 2657—2669.
Shin, D.-Y., Chung, J., Joe, Y., & others. (2012). Pretreatment with CO-releasing molecules suppresses hepcidin expression during inflammation and endoplasmic reticulum stress through inhibition of the STAT3 and CREBH pathways. Blood, 119(11), 2523—2532.
Silva-Sanchez, A., & Randall, T. D. (2020). Anatomical Uniqueness of the Mucosal Immune System (GALT, NALT, iBALT) for the Induction and Regulation of Mucosal Immunity and Tolerance. Mucosal Vaccines, 21.
Souquette, A., & Thomas, P. G. (2018). Past Life and Future Effects—How Heterologous Infections Alter Immunity to Influenza Viruses. Frontiers in immunology, 9,
Sousa, F. L., Thiergart, T., Landan, G., Nelson-Sathi, S., Pereira, I. A. C., & Allen, J. F., et al. (2013). Early bioenergetic evolution. Philosophical Transactions of the Royal Society B: Biological Sciences, 368(1622).
Uda, S. (2020). Application of information theory in systems biology. Biophysical Reviews, 12(2), 377.
Vestbo, J., Anderson, W., Coxson, H. O., & others. (2008). Evaluation of COPD Longitudinally to Identify Predictive Surrogate End-points (ECLIPSE). Eur. Respir. J. 31(4), 869—873.
Wagner, A., & Fell, D. A. (2001). The small world inside large metabolic networks. Proc. R. Soc. Lond. B Biol. Sci. 268(1478), 1803—1810.
Wang, M., & Kaufman, R. J. (2016). Protein misfolding in the endoplasmic reticulum as a conduit to human disease. Nature, 529(7586), 326—335.
Watve, M. G., & Yajnik, C. S. (2007). Evolutionary origins of insulin resistance: a behavioral switch hypothesis. BMC Evol. Biol. 7(1), 1—13.
West, N. R., McCuaig, S., Franchini, F., & Powrie, F. (2015). Emerging cytokine networks in colorectal cancer. Nat. Rev. Immunol. 15(10), 615—629.
Wilson, D. F. (2013). Regulation of cellular metabolism: programming and maintaining metabolic homeostasis. J. Appl. Physiol.
Wilson, D. F. (2015). Programming and regulation of metabolic homeostasis. American Journal of Physiology-Endocrinology and Metabolism,
Wilson, D. F. (2017). Oxidative phosphorylation: regulation and role in cellular and tissue metabolism. J. Physiol. 595(23), 7023—7038.
Ytting, C. K., Fuglsang, A. T., Kalervo Hiltunen, J., Kastaniotis, A. J., Özalp, V. C., & Nielsen, L. J., et al. (2012). Measurements of intracellular ATP provide new insight into the regulation of glycolysis in the yeast Saccharomyces cerevisiae. Integrative Biology, 4(1), 99—107.
Zhang, K., & Kaufman, R. J. (2008). From endoplasmic-reticulum stress to the inflammatory response. Nature, 454(7203), 455.
Zhang, C., Wang, G., Zheng, Z., & others. (2012). Endoplasmic reticulum-tethered transcription factor cAMP responsive element-binding protein, hepatocyte specific, regulates hepatic lipogenesis, fatty acid oxidation, and lipolysis upon metabolic stress in mice. Hepatology, 55(4), 1070—1082.

Deja una respuesta