Resistencia natural a la infección que funge acorde a cada reto específico y por demanda con protección del parénquima
La reciente publicación científica en Discover Medicine(1) sobre la respuesta celular constitutiva en resolver lesiones y la abrumadora defensa a la infección que acompaña a los procesos regenerativos comprendidos dentro dicha respuesta celular autónoma (no dependiente de células injmunitarias), encuentra ulterior validación por descubrimientos recientes que esclarecen la poderosa defensa que se ejerce dentro de esa ventana para lograr el dominio natural de la infección.
Por décadas se creyó que la función primaria de los péptidos derivados de la degradación del proteasoma, fuese la presentación de antígeno mediante el complejo de histocompatibildad mayor clase I para lanzar la inmunidad adaptativa por células T. Pero investigaciones durante la última década, han esclarecido que la función se extiende a la generación de péptidos antimicrobianos con propiedades catiónicas a escala y variedad inimaginable, mediante ulterior degradación tríptica protesómica provocados por cambios en la composición y función proteómica e inducida por patógenos para eventualmente inducir la extracción de dichas fracciones catiónicas, y que agujerean la membrana del patógeno, para causar el arresto del crecimiento bacteriano; sin participación adaptativa, y dentro todavía de la primera línea de defensa(2).
En 2011 Ponpuak y Deretic(3) describieron que en la autofagia los fagosomas eran más que una vacuola de residencia para la M. tuberculosis, dado que la molécula adaptadora p62 o secuestosoma 1, interactúa a la vez con substratos fagosómicos y secuestra componentes citosólicos inocuos (la proteína ribosómica rpS30, el precursor FAU y la ubiquitina) para procesarlos por degradación y liberar péptidos antimicrobianos obtenidos de material usado o a ser descartado. Esto le otorga al fagosoma cualidades lisosómicas y de maduración, que junto con la captura de hidrolasas y substratos intermedios de movilización y su acidificación, eventualmente se funden en un autolisosoma verdadero y propio. La suposición de que el producto de dicha transformación fuese simplemente aminoácidos libres a ser reciclados en momentos de inanición cambia. Desde entonces ha sido dilucidado que la proteólisis de estas proteínas citosólicas genera una mixtura de péptidos crípticos con poder antimicrobiano y que constituyen los nuevos antibióticos naturales llamados “críptidos” y que son generados de material biológico de descarte. En suma, la transformación potencia una pos-función muy valiosa, como la de eliminar la micobacteria, conocida por su formidable resistencia a ser dispuesta de ella; ver Fig. n°1.
Fig. 1
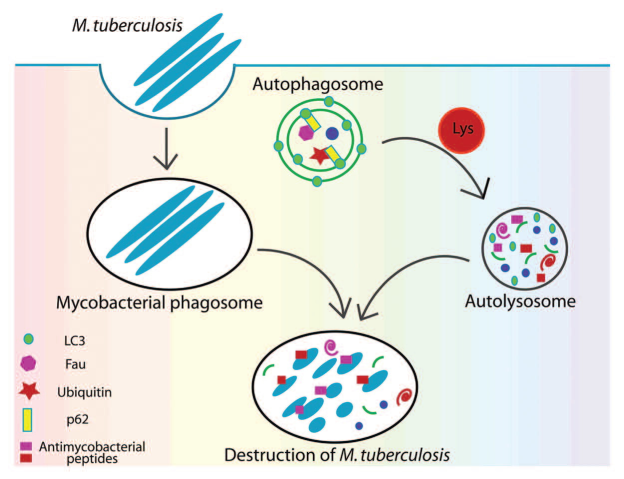
Eliminación de M. tuberculosis mediante autofagia y p62. La micobacteria viene fagocitada por macrófagos y mínimo por un tiempo reside dentro del fagosoma. Cuando se induce la autofagia, p62, como agente bifuncional, interactúa con substratos autofágicos y con LC3, para también reclutar substratos citosólicos. La maduración del autofagosoma y la captura de hidrolasa inducen la degradación de todos los substratos y su conversión en péptidos (criptidos) con poder antimicrobiano.
En marzo de 2025, Goldberg et al.(2) aclaran que la producción de criptidos con propiedades catiónicas también existe en la función proteasómica, la cual por décadas se creyó que se limitaba a generar el complejo de histocompatibilidad I o la degradación de proteínas inhibitorias (IkBs) para activar el factor nuclear kappa B (NF-kB) o rendir activos a precurssores proteícos con función reguladora transcripcional como, Spt23p, Mga2p y Epe1, pero que la fuente de extracción también se extiende a substratos extraídos del citosol, como en la autofagia. Además de ser un paso evolutivo de primer orden y que los péptidos antibacterianos suelen hallarse en una vasta variedad de tejidos y células, poseen un amplio espectro contra bacterias, hongos, virus y algunos parásitos. Además de su acción catiónica, también modulan la acción transcripcional y traslacional del hospedero. Han sido denominados como péptidos derivados del proteasoma (PDDPs p.s.e.i.) por Goldberg et al. Son constitutivos, pero también existe la posibilidad de inducir un aumento de su producción de cara a infecciones. Una unidad reguladora del proteasoma PSME3 promueve la degradación tríptica y esto aumenta la cantidad de los péptidos con terminales catiónicas. Análisis computacionales del fenómeno muestran que la mayoría de las proteínas del proteasoma están sujetas a dicha degradación y que, por tanto, el potencial de producción de PDDPs en el proteasoma es muy extenso. Una identificación in silico con el algoritmo pepsickle(4) de sobre posibles sitios de partición para obtener aminoácidos entre 10 a 50 unidades en longitud y calificados con un rango mínimo de eficacia basado en comparación estructural a péptidos con poder antibacteriano comprobado, señaló 270872 putativos con terminales carboxilicas catiónicas. Visto de otro ángulo, resulta que un 92% de los genes anotados poseen por lo menos un péptido con terminal catiónica. Lo que ilustra el enorme potencial que conlleva incitar la inmunidad celular autónoma para tratar lesiones tisulares. sin problemas de infección y de dar con la probabilidad contundente de que se genere el antimicrobiano a medida para combatir los patógenos involucrados según el caso específico.
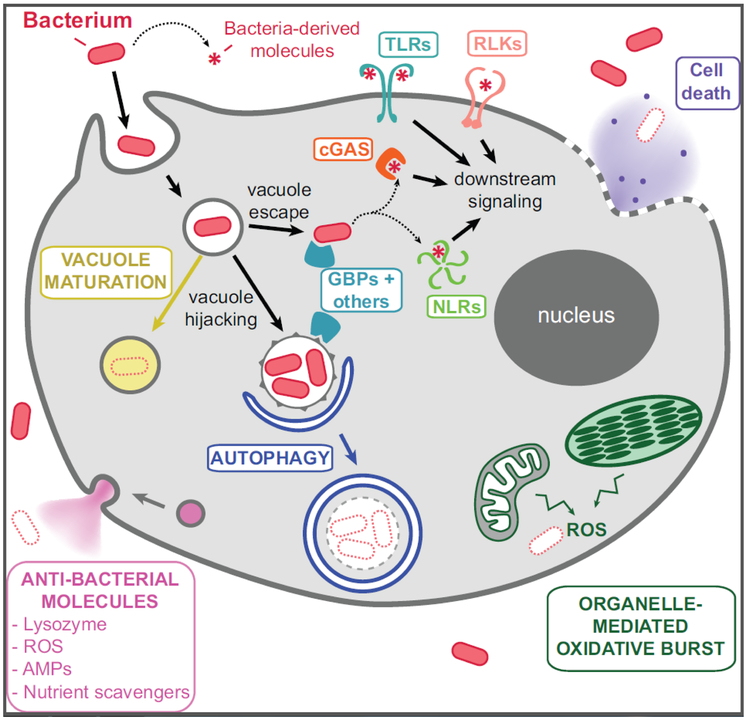
La fig 2 resume los mecanismos extra e intracelulas de los eucariotes para eliminar patógenos
La identificación del mecanismo de acción de Cubrisoft dentro de la inmunidad celular autónoma y la regular ausencia de infección en las lesiones tratadas con dicho producto concuerda con las investigaciones descritas en esta entrega. Tal evidencia coloca a Cubrisoft a la vanguardia en combatir la infección en tejidos lesionados, sin efectos tóxicos o no deseados y crea condiciones óptimas para la reparación regenerativa descrita en: The cell’s autonomous response window for advanced healing(1).
No es solo resistencia, sino también el manejo de la tolerancia, para preservar la salud de los tejidos y sobrevivir
Como se propone en el artículo, la reparación con fibrosis es algo de segundo orden, que no se valoró realmente hasta que se conoció la reprogramación celular. La creciente investigación desde el descubrimiento ha revelado múltiples formas dentro de la inmunidad innata mediante las cuales se evita la inflamación, lo que resulta en regeneración y ausencia de fibrosis. Y justo en el límite, aún se pueden encontrar varias opciones para sortear un microambiente con una ECM rígida y la consiguiente fuga de células inmunitarias.
Tampoco es sostenible afirmar que la fuga inmunitaria elimine el patógeno y elimine la infección, ya que los hallazgos clínicos del artículo y las grandes oportunidades que ofrecen los AMP para mantener las lesiones limpias son ahora un hecho, pero también por los refinamientos recientemente descubiertos de cómo funcionan las células asesinas naturales (NK) y las células similares a las células asesinas naturales (NK-like), tanto en la eliminación de patógenos como en el mantenimiento de la salud tisular. Es decir, una línea principal de linfocitos circulantes/residentes en el tejido asegura un papel de resistencia/tolerancia antes de dar paso a la inmunidad adaptativa y evita daños tisulares con un sorprendente parecido a cómo los intrusos son destrozados por los AMP, pero junto con la producción de anfiregulina. No solo eso, sino que también se han identificado amplias oportunidades en cuanto a la reprogramación del tejido pulmonar para la restauración de la salud, sin obstáculo al intercambio de gases, como resultado preferido, todas asequibles cuando se busca el control del tejido/microambiente (la principal forma de comunicarse con el interactoma). (6) (7) (8)
Existe una serie de respuestas celulares y sistémicas que protegen el parénquima del estrés, la disfunción y/o el daño tisular con miras a contener la severidad de las enfermedades y que no se relaciona con la severidad de la carga patógena. Son en gran medida adaptaciones metabólicas y redox de tipo antioxidante, que se ocupan de limitar el daño, y en esencia, manetener la salud del tejido/microambiente. (9)
En otras palabras, la descripción del artículo sobre la reparación dentro de la ventana autónoma necesita una mayor clarificación sobre cómo se restaura la arquitectura tisular cuando el método se utiliza inmediatamente luego de sufrir una lesión. Sin embargo, es un tema para un artículo futuro. Por favor, considerad esta sección final del comentario como un avance sobre el equilibrio requerido para combatir una lesión y lo que está por venir en el nuevo artículo, que oscila entre resistencia y tolerancia para lograr regeneración (futura práctica clínica estándar) mediante la reprogramación celular o paligenosis, y que exquisitamente evita el daño tisular durante el proceso de reparación. Finalmente, tomad en cuenta que estos mecanismos innovadores son características fundamentales de la inmunidad innata, y es sorprendente cómo todo esto haya pasado inadvertido durante tanto tiempo.
Referencias
1. Feoli-Tufi E, Redondo GM, Flores JS, Jara DP. The cell’s autonomous response window for advanced healing. Discov Med [Internet]. 10 de octubre de 2025 [citado 9 de noviembre de 2025];2(1):1-42. Disponible en: https://link.springer.com/article/10.1007/s44337-025-00478-4
2. Goldberg K, Lobov A, Antonello P, Shmueli MD, Yakir I, Weizman T, et al. Cell-autonomous innate immunity by proteasome-derived defence peptides. Nature [Internet]. marzo de 2025 [citado 9 de noviembre de 2025];639(8056):1032-41. Disponible en: https://www.nature.com/articles/s41586-025-08615-w
3. Ponpuak M, Deretic V. Autophagy and p62/sequestosome 1 generate neo-antimicrobial peptides (cryptides) from cytosolic proteins. Autophagy [Internet]. 1 de marzo de 2011 [citado 9 de noviembre de 2025];7(3):336-7. Disponible en: https://doi.org/10.4161/auto.7.3.14500
4. Weeder BR, Wood MA, Li E, Nellore A, Thompson RF. pepsickle rapidly and accurately predicts proteasomal cleavage sites for improved neoantigen identification. Bioinformatics [Internet]. septiembre de 2021;37(21):3723-33. Disponible en: https://doi.org/10.1093/bioinformatics/btab628
5. Richter Daniel J, Levin Tera C. The origin and evolution of cell-intrinsic antibacterial defenses in eukaryotes. Current opinion in genetics \& development 2019; 58-59:111–122.
https://pmc.ncbi.nlm.nih.gov/articles/PMC9275444/#F2
6. Schuster, I. S., Sng, X. Y. X., & Lau, C. M. (2023). La infección induce células NK de memoria residentes en el tejido que salvaguardan la salud del tejido. Inmunidad, 56(3), 531–546.
7. Vick, S. C., Domenjo-Vila, E., & Frutoso, M. (2025). Las células NK del tejido mucoso ajustan su función entre la actividad óptima antipatógeno y la protección tisular. bioRxiv.
8. Basil, M. C., Alysandratos, K.-D., & Kotton, D. N. (2024). Reparación y regeneración pulmonar: modelos avanzados y conocimientos sobre enfermedades humanas. Célula madre celular, 31(4), 439–454.
9. Soares, M. P., Gozzelino, R., & Weis, S. (2014). Tissue damage control in disease tolerance. Trends in Immunology, 35(10), 483–494.

Deja una respuesta